原始的な中央代謝系で働く酸化還元酵素の立体構造
- 発表者
-
Yan Zhen(東京大学大学院農学生命科学研究科 博士課程3年;当時)
丸山 茜(東京大学大学院農学生命科学研究科 修士課程2年;当時)
荒川 孝俊(東京大学大学院農学生命科学研究科 助教)
伏信 進矢(東京大学大学院農学生命科学研究科 教授)
若木 高善(東京大学大学院農学生命科学研究科 教授;当時)
発表のポイント
◆原始的な古細菌の中央代謝系で働く2-オキソ酸:フェレドキシン酸化還元酵素(OFOR)の最も単純な種類の結晶構造を決定し構造機能相関を解析した。
◆OFORの中で最も単純なドメイン構成でありながら多様な基質特異性を示す酵素の構造・機能との相関を初めて明らかにした。
◆本成果は、生命の起源に関わる原始的な古細菌の代謝の理解という基礎方面や、中央代謝系においてOFORが働いている病原性微生物の駆除という応用方面につながる重要な研究成果であるといえる。
発表概要
生物の栄養源であるグルコースは細胞内で分解されて、ピルビン酸やケトグルタル酸になりますが、これらの2−オキソ酸は、CoA(補酵素A)と反応して炭酸ガスとアシルCoAとに変換され、同時に電子伝達体となる補因子が還元されます。
このとき電子を受け取るのにフェレドキシンというタンパク質を用いる場合があります。少数の原始的な生物やすべての超好熱菌・古細菌では、2−オキソ酸:フェレドキシン酸化還元酵素(OFOR)がこの反応を可逆的に触媒します。
ピルビン酸専用のOFORであるピルビン酸:フェレドキシン酸化還元酵素(PFOR)と異なり、超好熱性古細菌(注1)スルホロバスでは、同一の酵素(StOFOR)がピルビン酸やケトグルタル酸の両方を用いることが出来るので、この酵素は中央代謝(注2)で重要な役割を担います。それにもかかわらずStOFORは4ドメイン(注3)からなり、PFORよりも構成が単純であることが知られていました。
今回、東京大学大学院農学生命科学研究科の若木高善前教授らの研究グループは、スルホロバスから2種類の酵素StOFOR1・StOFOR2を単離精製しX線結晶構造解析(注4)によって立体構造を決定することに成功しました。これによって、従来7ドメイン型に限られていたOFORの構造的な知見が広がり、外部の電子伝達体であるフェレドキシンとの相互作用についても新しい考察が可能となりました。
発表内容

図1 StOFOR、DaPFORのドメイン構成(a)と単量体の立体構造(b, StOFOR2; c, DaPFOR)。(拡大画像↗)
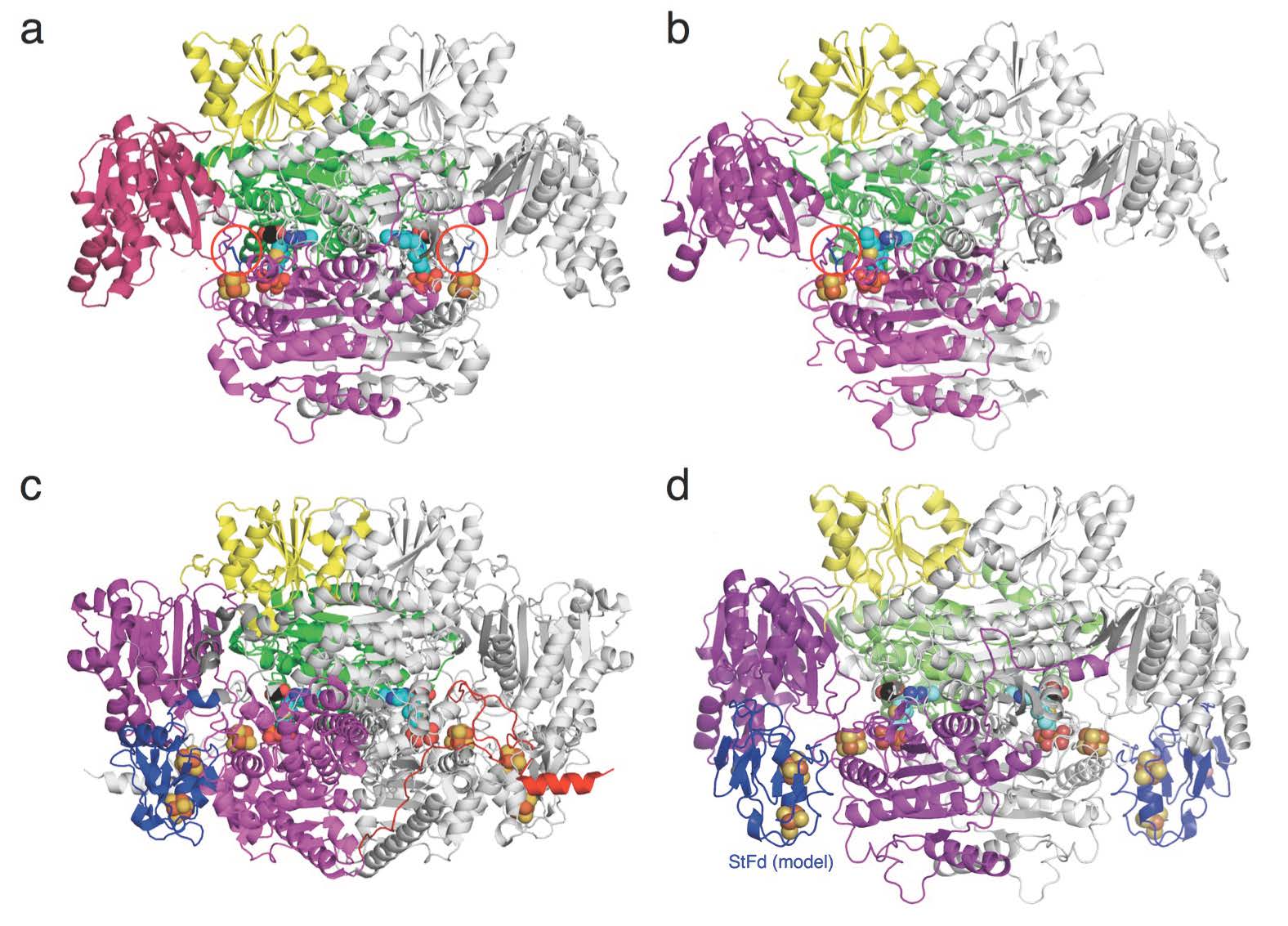
図2 a, StOFOR2; b, StOFOR1; c, DaPFOR; d, StOFOR2-StFd複合体(モデル)の全体構造。(拡大画像↗)
超好熱性古細菌の原始的な代謝系には他の生物には見られない特異な酵素が多数発見されています。若木高善前教授らの研究グループは、好酸好熱性古細菌スルホロバスから2-オキソ酸:フェレドキシン酸化還元酵素(StOFOR1)を1996年に単離精製し、これが分子量7万と3万の2種類のサブユニットからなり、基質である2-オキソ酸としてピルビン酸以外にもケトグルタル酸など広く利用できることから、中央代謝で重要な酵素であることを解明していました。しかし、唯一立体構造が報告された細菌型ピルビン酸:フェレドキシン酸化還元酵素(DaPFOR)が7個のドメインから構成されているのに対し、StOFORは4個のドメインしかなく、「分子内フェレドキシン」ドメインが欠損していました。さらに、StOFORは基質である2−オキソ酸に対する特異性が広く、中央代謝やアミノ酸代謝で重要な働きをするものと思われますが、その構造的な基盤は不明でした。
酵素の機能を理解するためには、その立体構造をX線結晶構造解析によって決定することが重要です。StOFOR1の結晶構造解析は難航しましたが、そのパラログ(注5)であるStOFOR2とともに構造決定に成功し、両者の立体構造が明らかになりました。図1aに、StOFORとPFORのドメイン構成の比較を、図1b、cに、今回報告したStOFOR2、DaPFORの立体構造をそれぞれ示します。
これらの3種類の酵素では、アミノ酸配列上はドメインの配置順序が異なる(図1a)にもかかわらず、全体的な立体構造の中心部分は類似していました(図1b, c)。実際には、これらの構造がそれぞれさらに対になり二量体になっていますが(図2)、StOFORにはドメインIV、V(分子内フェレドキシン)、VIIがないために、DaPFOR(図2c)と比べると、太ったミミズクが羽を広げたような形に見えます(図2a,b)。ところが、私たちが20年前に立体構造を決定したスルホロバス由来フェレドキシン(StFd)を、この羽の下にあてがうと、DaPFORのドメインVの位置にすっぽりはまって、全体構造がよく似てきます(図2dの青い領域)。
OFORは2-オキソ酸から外来のフェレドキシンに電子を2個渡しますが、StOFORではドメインVを介さずに直接StFdへと電子を伝達することがわかりました。
さらに、StOFORの2−オキソ酸結合部位はDaPFORと比べると、ピルビン酸に限らず、それよりも少し大きなケトグルタル酸も結合出来るだけの奥行きがあります。実際にStOFOR1, 2はピルビン酸とケトグルタル酸のどちらも基質としますが、活性中心で基質の結合にかかわるいくつかの残基を置換することで、たとえばケトグルタル酸とは結合できなくなることを示しました。
本成果は、生命の起源に関わる原始的な古細菌の中央代謝系で働く鍵となる酸化還元酵素の解明という基礎方面や、中央代謝系においてOFORが働いているトリコモナス、ジアルジア、ピロリ菌などの病原性微生物を駆除するためのOFOR阻害剤の設計という応用方面につながる重要な研究成果であるといえます。
発表雑誌
- 雑誌名
- 「Scientific Reports」
- 論文タイトル
- Crystal structures of archaeal 2-oxoacid:ferredoxin oxidoreductases from Sulfolobus tokodaii
- 著者
- Zhen Yan, Akane Maruyama, Takatoshi Arakawa, Shinya Fushinobu & Takayoshi Wakagi*
- DOI番号
- 10.1038/srep33061
- 論文URL
- http://www.nature.com/articles/srep33061
問い合わせ先
東京大学大学院農学生命科学研究科 応用生命工学専攻 酵素学研究室
教授 伏信 進矢
Tel:03-5841-5151
Fax:03-5841-5151
研究室URL:http://enzyme13.bt.a.u-tokyo.ac.jp/
用語解説
- 注1 超好熱性古細菌(hyperthermophilic archaea)
- 生育至適温度が80˚C以上の超好熱菌の大部分を占める古細菌(真核生物Eukaryaや真正細菌Bacteriaとは分類上異なる微生物群)で、生命の起源に関わる原始的な代謝系をもつと考えられる。
- 注2 中央代謝(central metabolism)
- 糖、アミノ酸、脂肪酸に関連した異化同化の両反応およびそれらに関するエネルギー代謝を中央代謝あるいは一次代謝という。
- 注3 ドメイン(domain)
- タンパク質の立体構造はいくつかの部分的なまとまり(ドメイン)に分けることが出来る。タンパク質は様々なドメインの組み合わせで進化したと考えられる。
- 注4 X線結晶構造解析(X-ray crystallography)
- 酵素を含むタンパク質の立体構造を明らかにするための最も一般的な方法の一つ。目的物質の結晶にX線を照射し、回折データを測定することにより、微細な三次元構造を知ることができる。
- 注5 パラログ(paralog)
- 同一生物のゲノム内で遺伝子重複によって生じた類似遺伝子またはその産物。