制限酵素が目的配列を探索する際の新規なメカニズム
- 発表者
-
王 徳龍(東京大学大学院農学生命科学研究科 応用生命化学専攻 博士課程2年)
宮園 健一(東京大学大学院農学生命科学研究科 応用生命化学専攻 特任助教)
田之倉 優(東京大学大学院農学生命科学研究科 応用生命化学専攻 教授)
発表のポイント
◆DNAグリコシラーゼ活性によって作用する制限酵素R.PabIが、認識配列でない二本鎖DNAと結合した状態の立体構造を決定しました。
◆R.PabIが二本鎖DNA上で形成するリング状の構造は、効率的なDNA分解に必要であることを明らかにしました。
◆生命現象の根幹となる、DNA結合タンパク質による認識配列探索機構の一端を解明できました。
発表概要
制限酵素は、特定の塩基配列を認識し二本鎖DNAの分解を行う酵素群です。制限酵素が効率よくDNAの分解を行うためには、その制限酵素の認識配列を、それ以外の大多数の塩基配列の中から効率よく見つけ出す必要があります。
今回、東京大学大学院農学生命科学研究科の田之倉優教授を中心とする研究グループは、X線結晶構造解析法(注1)とさまざまな分析手法を組み合わせることにより、超好熱古細菌(注2)Pyrococcus abyssi由来の制限酵素R.PabIが、どのような機構を用いて認識配列を見つけ出すかを解析しました。R.PabIは既知の制限酵素とは異なり、二本鎖DNAを囲むリング状の構造で、認識配列でない二本鎖DNAと結合します。このリング状の構造は、R.PabIが効率よく認識配列を見つけだす上で重要な構造です。
今回明らかにしたR.PabIによる認識配列の探索機構は、既知のものとは異なる新規の機構であり、生命現象の根幹となるタンパク質-DNA間相互作用の新たな一面を明らかにすることができました。
発表内容
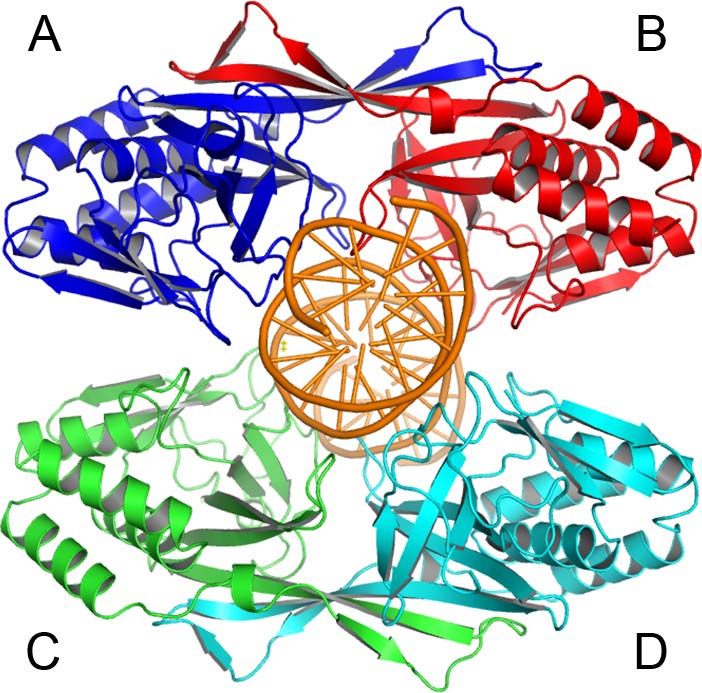
図1 X線結晶構造解析法により決定したR.PabIとその認識配列を含まないDNAの複合体構造
二つのR.PabI二量体(青・赤(AB)と緑・水色(CD)) はリング状の四量体構造をとり、認識配列以外の二本鎖DNA(オレンジ)と結合をします。
(拡大画像↗)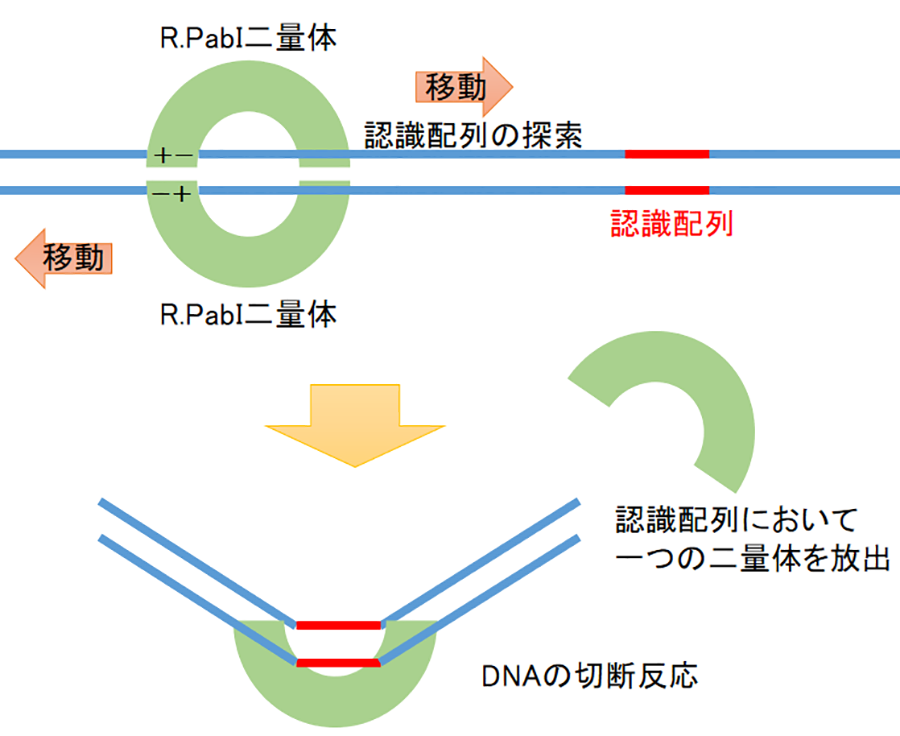
図2 予想されるR.PabIによる認識配列探索機構
(拡大画像↗)
細菌の外来DNAに対する自己防御機構にかかわるタンパク質として発見された制限酵素は、特定の塩基配列を認識して二本鎖DNAの切断反応を触媒する酵素です。自身のゲノムDNAを保護するために、制限酵素はターゲットとなる外来DNAを素早く認識して処理する必要があります。制限酵素は、特定の塩基配列を認識して二本鎖DNAの切断を行う酵素であるため、効率的なDNA切断のためには、大多数の「認識しない塩基配列」の中から、「特定の塩基配列」を素早く探しだす必要があります。一般的に、特定の塩基配列を認識するタンパク質は、その認識する塩基配列に直接結合するわけではなく、DNAの塩基配列に関係なく二本鎖DNAに結合した後、その位置を微調整しながら特定の配列を見つけ出すといわれています。制限酵素R.PabIは超好熱古細菌Pyrococcus abyssi由来の制限酵素であり、通常の制限酵素とは異なり、DNAグリコシラーゼ活性によってDNAの切断を行う非常に珍しい制限酵素です。本研究グループは、R.PabIがどのような機構で認識する塩基配列を探索するかを、構造学的な手法により明らかにしようと試みました。
R.PabIと認識配列を含まない二本鎖DNAの複合体の立体構造をX線結晶構造解析法により決定するため、R.PabIと二本鎖DNAの複合体結晶を作製し、大型放射光施設(注3)Photon Factoryのタンパク質結晶構造解析ビームラインAR-NE3AにてX線回折データを収集しました。得られたX線回折データを用いてR.PabIと二本鎖DNAの複合体構造を決定したところ、二つのR.PabI二量体が二本鎖DNAを囲うようなリング状の構造をとることが明らかになりました(図1)。このリング状の構造は、正及び負の電荷をもつ二つのアミノ酸間の相互作用によって安定化されています。この分子間の相互作用がR.PabIの活性に及ぼす影響を、R.PabIの一部の構造に対し人工的に変異を導入したR.PabI変異体タンパク質を用いて調べたところ、リング状の構造を安定化させる残基は、R.PabIが効率よくDNAを切断するために重要であることが明らかになりました。構造解析の結果明らかになったR.PabIのリング状の構造は、R.PabIが大多数の認識配列でない塩基配列から、特定の塩基配列を探し出す際に重要であることが示唆されました。このような機構は、これまでの研究では明らかにされていない新規の機構です。R.PabIが認識配列を見つけた後は、このリング状の構造が解消され、DNAの切断のために必要な構造へと変化することが予想されます(図2)。
今回の研究では、DNAの塩基配列を読むことのできるタンパク質の一種である制限酵素が、膨大なDNA情報の中から特定の塩基配列を効率よく探し出す新たな機構を解明することができました。生命は、DNA上に記載された塩基配列の情報を基に成り立っています。そのため、その塩基配列の情報を読み取る際に必須なタンパク質-DNA間の相互作用様式の解明は、生命現象を理解する上で欠かせません。本成果は、タンパク質とDNAの相互作用機構の理解を深めるうえで重要な研究成果であるといえます。
本研究は、文部科学省「タンパク3000プロジェクト」、「ターゲットタンパク研究プログラム」、「創薬等支援技術プラットフォーム事業」及び、日本学術振興会科学研究費助成事業の支援を受けて行われました。
発表雑誌
- 雑誌名
- 「Scientific Reports」
- 論文タイトル
- Tetrameric structure of the restriction DNA glycosylase R.PabI in complex with nonspecific double-stranded DNA
- 著者
- Delong Wang, Ken-ichi Miyazono, and Masaru Tanokura*
- DOI番号
- 10.1038/srep35197
- 論文URL
- http://www.nature.com/articles/srep35197
問い合わせ先
<研究に関すること>
東京大学大学院農学生命科学研究科 応用生命化学専攻 食品生物構造学研究室
教授 田之倉 優
Tel:03-5841-5165
Fax:03-5841-8023
E-mail:amtanok@mail.ecc.u-tokyo.ac.jp
用語解説
- 注1 X線結晶構造解析法
- 生体高分子の結晶を作製しそこにX線を照射すると、その結晶に特徴的な回折像を得られます。この回折像は、結晶を形成する分子の構造情報と相関するため、そのパターンや強度を解析することによって、目的分子の立体構造を決定することができます。
- 注2 超好熱古細菌
- 温泉や熱水域、強く発酵した堆肥、熱水噴出孔などの高温環境を好む古細菌で、80℃以上の環境で生育します。
- 注3 大型放射光施設
- タンパク質の立体構造解析の精度は、構造解析の際に用いるX線回折データの品質(分解能)に大きく依存します。正確な立体構造を決定するためには、高品質・高強度なX 線を利用できる大型放射高施設の利用が必須です。