蛾の性フェロモン生合成を司るペプチドの結合に重要な受容体の部位を特定
- 発表者
-
河合 岳志(東京大学大学院農学生命科学研究科 応用生命化学専攻 特任研究員;当時)
片山 幸江(東京大学大学院農学生命科学研究科 応用生命化学専攻 特任講師)
郭 琳珺(東京大学大学院農学生命科学研究科 応用生命化学専攻 博士課程3年)
刘 德生(東京大学大学院農学生命科学研究科 応用生命化学専攻 博士課程3年)
鈴木 達也(東京大学大学院農学生命科学研究科 応用生命化学専攻 修士課程2年;当時)
早川 江(東京大学大学院農学生命科学研究科 応用生命化学専攻 特任研究員;当時)
長澤 寛道(東京大学大学院農学生命科学研究科 応用生命化学専攻 教授;当時)
田之倉 優(東京大学大学院農学生命科学研究科 応用生命化学専攻 教授)
永田 宏次(東京大学大学院農学生命科学研究科 応用生命化学専攻 准教授)
発表のポイント
◆蛾の性フェロモンの生合成を司る神経ペプチド(PBAN)と結合するPBAN受容体の機能に重要なアミノ酸残基を特定し、PBANとPBAN受容体が結合した状態のモデルを構築しました。
◆PBAN受容体で特定した重要なアミノ酸残基の多くは、アミノ酸配列がPBAN受容体に類似しているヒトのニューロメジンU受容体でも存在していました。
◆人為的に蛾の性フェロモンを生合成して害虫を抑制する技術の開発だけでなく、ヒトのストレスや食欲等の制御に関わるニューロメジンUの作用機構の理解にも役立つと期待されます。
発表概要
蛾の雌が交配のため性フェロモンを利用して、雄を誘き寄せていることは広く知られています。雌の蛾の体内では、頭部にある神経細胞からフェロモン生合成活性化神経ペプチド(pheromone biosynthesis-activating neuropeptide, PBAN)と呼ばれる33個のアミノ酸らなる神経ペプチド(注1)が分泌され、腹部末端のフェロモン腺にあるPBAN受容体にPBANが結合することで、初めて性フェロモンが作られます。しかし、PBAN とPBAN受容体の結合様式やPBAN受容体の機能に重要なアミノ酸残基(注2)は未解明のままでした。
東京大学大学院農学生命科学研究科の田之倉優教授、永田宏次准教授らの研究グループは、PBAN受容体の①細胞膜への移行、②PBANへの結合、③PBANとの結合を細胞内に伝える情報伝達、という3つの機能に重要なアミノ酸残基をPBAN受容体の点変異体解析(注3)により明らかにしました(図1)。また、コンピュータシュミレーションによって上記の解析データをうまく説明できるPBANとPBAN受容体が結合した複合体の立体構造モデルを構築しました(図2)。同時に、PBANと活性部位のアミノ酸配列が類似しているヒトの神経ペプチド、ニューロメジンU(NMU)とその受容体の複合体の立体構造モデルも構築し、PBANとNMUの活性部位が類似の様式で各受容体に結合することを提唱しました。
本研究により同定されたPBAN受容体の機能に重要なアミノ酸残基の位置は、既に結晶構造が明らかになっている低分子化合物を認識するGタンパク質共役受容体(GPCR、アデノシン受容体やβアドレナリン受容体、注4)の重要アミノ酸残基の位置とよく対応していましたが、一方でPBAN受容体とNMU受容体に特徴的な重要残基も見出されました。この成果は、PBANとPBAN受容体の結合を阻害する害虫防除剤の開発に役立つだけでなく、ヒトの摂食抑制作用、ストレス反応の調節、痛みの制御などに関わっているNMUの受容体認識機構についても新たな情報を提供するものです。
発表内容
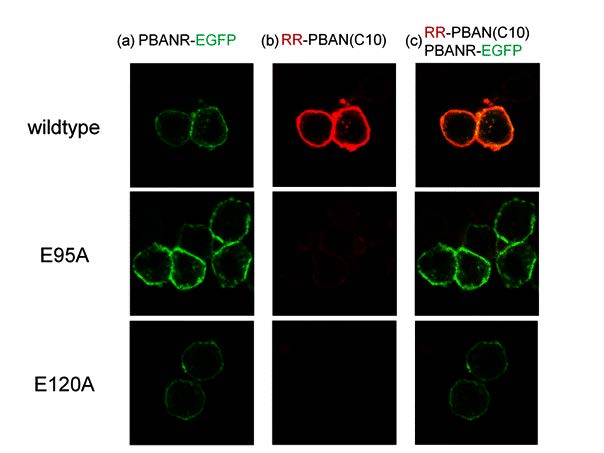
図1 PBAN受容体の点変異体解析。変異のない天然型(wildtype)と変異体2種(E95、E120)のデータを示す。(a)昆虫細胞に発現させたPBANR-EGFP由来の緑色蛍光を検出、(b) PBANR-EGFPに結合したRhodamine Red (RR)-PBAN(C10)(PBAN C末端部10残基断片)由来の赤色蛍光を検出、(c) PBANR-EGFP由来の緑色蛍光とRR-PBAN(C10)由来の赤色蛍光とを重ね合わせた像。天然型PBANR-EGFPが細胞膜上に分布しRR-PBAN(C10)と強く結合するのに対し、変異体2種は細胞膜上に分布するもののRR-PBAN(C10)との結合が弱いことが示された。(拡大画像↗)
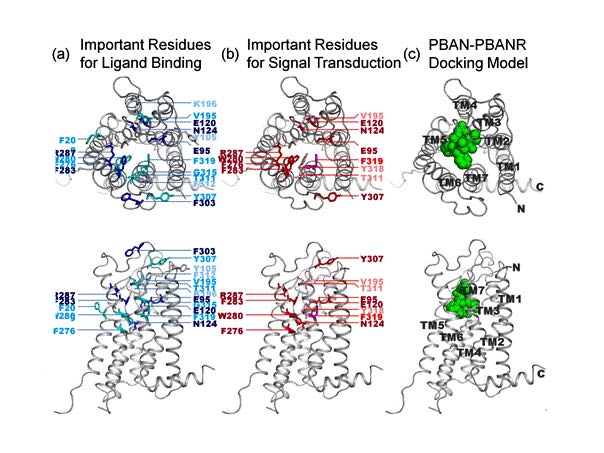
図2 PBAN受容体の点変異体解析により明らかとなった (a) PBANとの結合に重要な残基と (b) 情報伝達に重要な残基。(c) ドッキングシミュレーションにより得られたPBAN C末端活性部位とPBAN受容体複合体の構造モデル。PBAN C末端活性部位を緑の球体で示す。PBAN受容体は7本の細胞膜貫通へリックスを有しており、N末端から順にTM1-TM7と呼ばれる((c)中に図示)。複合体構造モデルにおいて、PBAN C末端活性部位がPBAN受容体の重要残基に囲まれているため、このモデルは大きくは間違っていないと期待されるが、立体構造に基づく薬剤設計(structure-based drug design)を行うためにはX線結晶構造解析により複合体結晶構造を解くことが必要である。(拡大画像↗)
雌の蛾は、成虫になった後、頭部の神経節から性フェロモン生合成を活性化する33残基の神経ペプチド(PBAN)を体液中に分泌し、PBANが尾部末端の性フェロモン産生器官であるフェロモン腺のPBAN受容体(PBANR)に結合することで初めて性フェロモンが生合成されます。PBANはC末端部にFX1PRX2-NH2モチーフ(X1、X2は任意のアミノ酸残基、NH2はC末端カルボキシル基がアミド化されていることを示す、注5)を有し、このC末端5残基と末端アミド基(FSPRL-NH2)が活性に必須であること、またPBANRはロドプシン様Gタンパク質共役受容体(GPCR)の一種であることが示されていました。しかし、PBANとPBANRの結合様式やPBANRの機能に重要なアミノ酸残基は未解明のままでした。
東京大学大学院農学生命科学研究科の田之倉優教授、永田宏次准教授らの研究グループは、PBANRのPBAN認識に関与する可能性のあるアミノ酸残基27個を1個ずつアラニン残基に置換した点変異体27種について、C末端側に緑色蛍光タンパク(EGFP)を付加したPBANR変異体-EGFP融合タンパク質を昆虫細胞に発現させ、この融合タンパク質の①発現量および膜への移行能、②PBANへの結合能、③PBANとの結合を細胞内に伝える情報伝達能、の3つの機能をそれぞれ調べました。①ではEGFP緑色蛍光の強度からPBANR変異体-EGFPの発現量およびその局在を観察して、②では蛍光色素Rhodamine Redで標識したPBAN C末端10残基を細胞外に添加し、洗浄後細胞上のEGFPとRhodamine Redの蛍光強度を計測し、蛍光強度比から各PBANR変異体のPBANへの結合能を求めました(図1)。③ではPBANとの結合によりPBANRが活性化されると細胞内カルシウム濃度が上昇することが知られているため、カルシウムの濃度変化に蛍光を発生する試薬を用いてPBANR変異体-EGFP発現細胞にPBAN C末端10残基を加えた際の細胞質内カルシウムイオン濃度の変動を調べました。こうして特定した重要残基のうち、細胞膜への移行に重要な4アミノ酸残基は細胞膜貫通へリックス5, 6(図2(c)のTM5, 6)上に位置していました。一方、PBANとの結合に重要なアミノ酸残基と情報伝達に重要なアミノ酸残基は大部分一致しており、それらは細胞膜貫通へリックス2, 3, 6, 7(図2(c)のTM2, 3, 6, 7)上に位置していました。このことから、PBANRにおけるPBANとの結合と細胞内への情報伝達との連携が実験的に確かめられました。他方で、PBANRのホモロジーモデリング(注6)およびPBANとのドッキングシミュレーション(注7)から、上記変異体解析の結果を良く説明できるPBANとPBANRのドッキングモデルを構築することに成功しました(図2)。同時並行で、PBANとPBANRのオルソログ(注8)であるヒトのニューロメジンU(NMU)とニューロメジンU受容体(NMUR)についてもドッキングモデルを構築し、PBANとNMUの活性に重要な共通モチーフ(FX1PRX2-NH2モチーフ。PBANではFSPRL-NH2、NMUではFRPRN-NH2)が各々の受容体に似た様式で結合することを提唱しました。特にPBANとNMUの活性に最も重要なC末端のアミノ酸残基と末端アミドの認識に、膜貫通へリックスIIとIII上に位置する2個のグルタミン酸残基が関与することをドッキングモデルに基づいて提唱しました。この2個のグルタミン酸残基はPBANRとNMURに特徴的で、他のGPCRでは保存されていません。X線結晶構造によってすでに構造が明らかになっているA2aアデノシン受容体(A2aAR)やβ2アドレナリン受容体(β2AR)との構造比較から、PBANRやNMURがPBANやNMUのC末端活性部位を認識すると推定される残基はA2aARやβ2ARのアゴニスト(注9)認識残基とほぼ対応する位置に分布していることが示されました。
近年、ロドプシン様GPCRのX線結晶構造解析が盛んに行われ、その立体構造やリガンドとの結合様式が、徐々に明らかになりつつあります。しかし、アゴニストとして作用する生理活性ペプチドがGPCRに結合した結晶構造は、まだニューロテンシン(NTS)とニューロテンシン受容体複合体の1例しか報告されておらず、NTS結合部位は今回提唱した推定PBAN結合部位とは位置が異なっています。このように神経ペプチドやペプチドホルモンとその受容体であるGPCRとの相互作用様式やGPCRの活性化機構については未解明な事柄が多く残されています。特にPBANのオルソログであるNMUはヒトの摂食抑制作用、ストレス反応の調節、痛みの制御などに関わっているため、本研究成果は、蛾の性フェロモン生合成の人為的制御による害虫防除を目的とした農薬の開発のみならず、医薬品開発の基盤情報となることが期待されます。
発表雑誌
- 雑誌名
- 「The Journal of Biological Chemistry」(289巻、2014 年、19150-19163ページ)
- 論文タイトル
- Identification of Functionally Important Residues of the Silkmoth Pheromone Biosynthesis-activating Neuropeptide Receptor, an Insect Ortholog of the Vertebrate Neuromedin U Receptor
- 著者
- Takeshi Kawai (河合岳志), Yukie Katayama (片山幸江), Linjun Guo (郭琳珺), Desheng Liu (刘德生), Tatsuya Suzuki (鈴木達也), Kou Hayakawa (早川江), Jae Min Lee (李載旼), Toshihiro Nagamine (永峰俊弘), J. Joe Hull, Shogo Matsumoto (松本正吾), Hiromichi Nagasawa (長澤寛道), Masaru Tanokura (田之倉優), and Koji Nagata (永田宏次)
- DOI番号
- 10.1074/jbc.M113.488999
- 論文URL
- http://www.jbc.org/content/289/27/19150.abstract
問い合わせ先
東京大学大学院農学生命科学研究科 応用生命化学専攻 食品生物構造学研究室
教授 田之倉 優
Tel:03-5841-5165
Fax:03-5841-8023
研究室URL:http://fesb.ch.a.u-tokyo.ac.jp//
准教授 永田 宏次
Tel:03-5841-2283
Fax:03-5841-2283
研究室URL:http://fesb.ch.a.u-tokyo.ac.jp//
用語解説
- 注1 神経ペプチド
- 神経末端から分泌され、情報伝達物質として働くペプチド(数個から数十個のアミノ酸が鎖状に縮合したもの)。C末端にFX1PRX2-NH2モチーフをもつ神経ペプチドとして、蛾のフェロモン生合成活性化神経ペプチド(PBAN)、ヒトのニューロメジンU(NMU)の他に、カイコガの休眠ホルモン、アワヨトウの体色黒化赤化ホルモン、バッタのミオトロピン、ハエのピロキニン等が知られている。これらの神経ペプチドは多様な生物活性を示す。
- 注2 アミノ酸残基
- タンパク質やペプチドは複数のアミノ酸が脱水縮合してペプチド結合を形成し鎖状につながったものである。このタンパク質やペプチド中の脱水縮合して繋がったアミノ酸のことをアミノ酸残基(または略して残基)と呼ぶ。アミノ酸の化学構造をNH2-CHR-COOH(ここでRはアミノ酸の種類により異なる部分で側鎖と呼ばれる)と表記する時、アミノ酸残基は-NH-CHR-CO-と表記される。「33個のアミノ酸からなるペプチド」を「33残基のペプチド」とも表現する。
- 注3 点変異体解析
- タンパク質の機能に重要なアミノ酸残基を特定するために、特定のアミノ酸残基を他のアミノ酸残基に置換して点変異体を作製し、その活性を変異のない天然型タンパク質と比較することにより、変異導入したアミノ酸残基の活性への寄与を評価する方法。本研究では、最も単純な側鎖構造をもつL型アミノ酸のL-アラニン残基に置換し、各アミノ酸残基側鎖の重要性を評価した。
- 注4 Gタンパク質共役受容体(GPCR)
- 細胞膜内に7回膜貫通ヘリックスドメイン、細胞外側から膜内にかけてリガンド結合部位、細胞内側にGタンパク質結合部位を有し、細胞外の化学的情報を細胞内に伝達する機能を有する膜タンパク質。GPCRは大きく3つのタイプに分類されるが、PBANRやNMURといった神経ペプチド受容体やβ2アドレナリン受容体(β2AR)といった生理活性アミン受容体が属するロドプシン様GPCRが大半を占める。細胞内のGタンパク質は生理的に不活性な状態ではGDPを結合したαドメインがβ、γの2つのドメインとヘテロ3量体を形成しており、受容体の活性化に伴ってGDP-GTP交換が生じてαドメインが解離すると共に受容体タンパク質からも解離する。現在使われている薬の約50%はGPCRに結合して作用する。
- 注5 モチーフ
- 各種タンパク質やペプチドに認められる特徴的なアミノ酸配列。機能や局所構造と関連づけるために用いられる場合が多い。
- 注6 ホモロジーモデリング
- コンピュータ上で、タンパク質の立体構造を予測する手法の一つ。立体構造が未知のタンパク質の構造を構築するために、まずそれにアミノ酸配列相同性があり立体構造が決定されているタンパク質を探索し、次にその立体構造を鋳型として構造未知のタンパク質の立体構造モデルを構築し、分子動力学法などにより構造を最適化する方法。
- 注7 ドッキングシュミレーション
- コンピュータ上で、リガンドと受容体タンパク質のように特異的に結合する2つの分子がどのように相互作用するのかを計算する手法。立体構造に基づく薬剤設計(structure-based drug design)にて利用される。
- 注8 オルソログ
- 共通の祖先遺伝子から生物種が分岐するのに伴って派生した複数の遺伝子(産物)の関係。
- 注9 アゴニスト
- 受容体に結合して生体内物質と同様の細胞内情報伝達系を作動させる化合物。アゴニストが受容体と結合すると受容体の立体構造変化が生じ、応答反応を引き起こす。