希少なオリゴ糖“β-1,2-グルコオリゴ糖”を細菌が捕まえるしくみを解明
- 発表者
-
阿部 紘一(東京大学 大学院農学生命科学研究科 応用生命工学専攻 博士課程学生(当時)/ 東京大学 大学院農学生命科学研究科 アグリバイオインフォマティクス教育研究ユニット 特任研究員(現在))
砂川 直輝(東京大学 大学院農学生命科学研究科 生物材料科学専攻 農学特定研究員)
寺田 透(東京大学 大学院情報学環・学際情報学府 / 東京大学 大学院農学生命科学研究科 アグリバイオインフォマティクス教育研究ユニット 准教授)
高橋 佑汰(新潟大学 大学院自然科学研究科 修士課程学生(当時))
荒川 孝俊(東京大学 大学院農学生命科学研究科 応用生命工学専攻 助教)
五十嵐 圭日子(東京大学 大学院農学生命科学研究科 生物材料科学専攻 准教授 / VTTフィンランド技術研究センター 客員教授)
鮫島 正浩(東京大学 大学院農学生命科学研究科 生物材料科学専攻 教授)
中井 博之(新潟大学 大学院自然科学研究科 准教授)
田口 速男(東京理科大学大学院 理工学研究科 教授)
中島 将博(東京理科大学大学院 理工学研究科 講師)
伏信 進矢(東京大学大学院 農学生命科学研究科 応用生命工学専攻 教授)
発表のポイント
◆β-1,2-グルコオリゴ糖を生物が取り込むしくみに関する初の知見が得られました。
◆細菌の感染に関わる糖質の生物代謝の詳細が判明しただけでなく、自然界で希少なオリゴ糖を検出する技術の開発に貢献することが期待されます。
発表概要
β-1,2-グルカン、およびそれよりも短いβ-1,2-グルコオリゴ糖は、澱粉やセルロースなどと同じくグルコース(ブドウ糖)から構成される糖質です。しかし、これらの糖質は入手困難であるために、その代謝に関わる酵素・タンパク質の研究例は極めて限られていました。近年、東京理科大学大学院理工学研究科の中島将博 講師らの研究グループによりβ-1,2-グルカンとβ-1,2-グルコオリゴ糖を合成・分解する酵素の働きが徐々に明らかになってきました。しかし、これらの糖質が生き物によってどのようにして捕捉され、細胞内に取り込まれるのかは不明でした。今回、東京大学大学院農学生命科学研究科の阿部紘一 博士課程学生(当時)、同研究科の伏信進矢 教授らの研究グループは、中島 講師らのグループと共同で研究を行い、細菌のβ-1,2-グルコオリゴ糖の取り込みに関する初の知見を得ることに成功しました。本研究の成果は、生物によるβ-1,2-グルコオリゴ糖の利用に関する謎を解き明かし、自然界で希少なβ-1,2-グルコオリゴ糖を検出するバイオセンサー(注1)の開発に繋がると期待されます。
発表内容
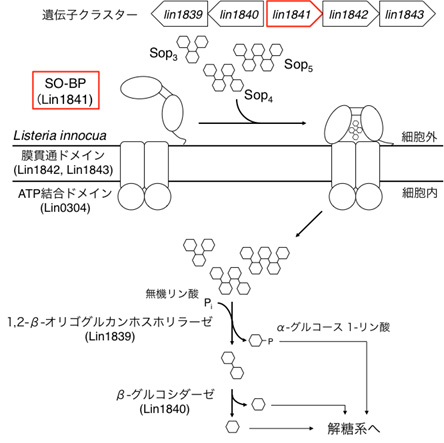
図1 細菌L. innocuaによるβ-1,2-グルカンおよびβ-1,2-グルコオリゴ糖の代謝経路
上にSO-BP(Lin1841)とその周辺の遺伝子を、下に先行研究と本研究で明らかになった代謝経路を示す。SO-BPは細胞表面でSop3〜Sop5を捕まえ、ABC輸送体(Lin1842、Lin1843、Lin0304)により細胞内に取り込む。取り込まれたβ-1,2-グルコオリゴ糖は、2種類の酵素(Lin1839とLin1840)によりグルコースとα-グルコース1-リン酸に分解され、細菌の栄養源となる。 (拡大画像↗)
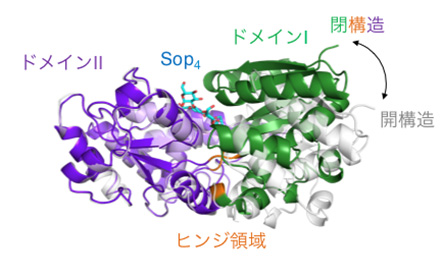
図2 SO-BPの全体構造とβ-1,2-グルコオリゴ糖の結合に伴う構造変化の様子(拡大画像↗)
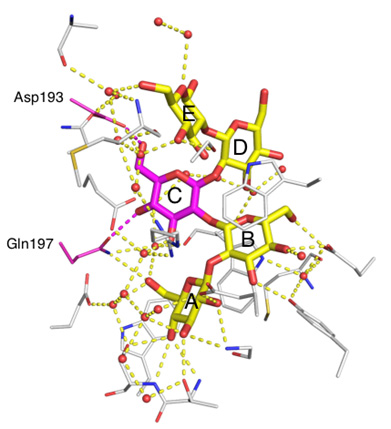
図3 SO-BPのβ-1,2-グルコオリゴ糖が結合する部分の構造
Sop5を太い棒、SO-BPのアミノ酸残基を細い棒、水分子を赤い球、水素結合を黄色の点線で表している。Sop5のグルコース単位を、奥に結合した部分から順にABCDEとラベルしている。Sop4とSop3も同様にしてA〜DまたはA〜Cの順に結合していた。SO-BPのβ-1,2-グルコオリゴ糖の結合に重要なアスパラギン酸残基(Asp193)、グルタミン残基(Gln197)、グルコース部分(C)を赤紫色で強調している。(拡大画像↗)
東京理科大学大学院理工学研究科の中島将博 講師らの研究グループは、以前よりβ-1,2-グルカンを合成・分解する酵素に着目して研究を行ってきました。近年、同研究グループは細菌Listeria innocuaからβ-1,2-グルカンおよびβ-1,2-グルコオリゴ糖を分解する2つの酵素、1,2-β-オリゴグルカンホスホリラーゼ(Lin1839)とβ-グルコシダーゼ(Lin1840)を発見し、その機能と立体構造を明らかにしました。これらの酵素遺伝子の隣にはATP結合カセット型(ABC)輸送体の溶質結合タンパク質(Lin1841)(注3)の遺伝子が存在します(図1)。このことからLin1841がβ-1,2-グルカンもしくはβ-1,2-グルコオリゴ糖の輸送に関わることが推測されました。しかし、実際にこれらの糖質がどのようにして捕捉され、輸送されるのかは不明でした。今回、東京大学大学院農学生命科学研究科 阿部紘一 博士課程学生(当時)と同大学院の伏信進矢 教授らのグループは、中島 講師のグループと共同で研究を行いました。
はじめに、等温滴定熱量測定(ITC)(注4)によりLin1841がどのような糖と結合するのかを調べたところ、Lin1841は3糖以上のβ-1,2-グルコオリゴ糖のみを選んで結合し、中でも重合度4前後の短いβ-1,2-グルコオリゴ糖を好むことが明らかとなりました。このことから、Lin1841をSopn結合タンパク質(SO-BP)と名付けました。次に、X線結晶構造解析(注5)により、SO-BPの立体構造を、糖が結合していない状態とSop3、Sop4、Sop5の3種のβ-1,2-グルコオリゴ糖が結合した状態の合計4状態で決定しました。糖が結合していない状態では、SO-BPの2つの大きなかたまりになっている部分(ドメインIとII)が大きく開いた構造をとっていたのに対し、3〜5糖との複合体構造では、2つのドメインの間にあるちょうつがいのような部分(ヒンジ領域)が折れたたんで閉じた構造をとっており、β-1,2-グルコオリゴ糖は2つのドメインに挟み込まれる形で結合していました(図2)。このことから、SO-BPはβ-1,2-グルコオリゴ糖の結合に伴い大きく構造を変えることが分かりました。また、結合したβ-1,2-グルコオリゴ糖は独特な“ジグザグ”構造をとっており、多数の相互作用により、SO-BPにしっかりと結合していました(図3)。この相互作用の様子はITCから推測されたものと良く一致しており、SO-BPが働く「かたち」と「しくみ」について、詳細かつ明確な知見が得られました。さらに、SO-BPが3糖以上のβ-1,2-グルコオリゴ糖を好んで結合する理由を分子動力学シミュレーション(注6)と部位特異的変異導入(注7)により調べたところ、Sop3の3つ目のグルコース部分(図3のCの部分)を結合している、193番目のアスパラギン酸残基(Asp193)と197番目のグルタミン残基(Gln197)が重要な役割を担っていることが明らかとなりました。これらの知見をふまえて、ゲノムデータベースを利用した分子系統学的な解析をおこなったところ、土壌細菌やビフィズス菌などの腸内細菌もSO-BPに類似した遺伝子を持っており、これらの細菌もβ-1,2-グルコオリゴ糖を取り込んで代謝している可能性が示されました。このことは、β-1,2-グルカンおよびβ-1,2-グルコオリゴ糖が、これまで考えられていたよりも自然界に普遍的に存在しているかもしれないことを意味しています。
本研究により、細菌がSO-BPを用いてβ-1,2-グルコオリゴ糖を捕らえる構造基盤が初めて明らかとなりました。本研究は生物によるβ-1,2-グルカンとβ-1,2-グルコオリゴ糖の代謝を考える上で重要な基礎的情報となり、新たな関連タンパク質の発見にも繋がると期待されます。また、SO-BPはβ-1,2-グルコオリゴ糖のみを強く結合することから、このような希少な糖質を感知するバイオセンサーとしての利用も期待できます。
発表雑誌
- 雑誌名
- :The Journal of Biological Chemistry
- 論文タイトル
- :Structural and thermodynamic insights into β-1,2-glucooligosaccharide capture by a solute-binding protein in Listeria innocua
- 著者
- :Koichi Abe, Naoki Sunagawa, Tohru Terada, Yuta Takahashi, Takatoshi Arakawa, Kiyohiko Igarashi, Masahiro Samejima, Hiroyuki Nakai, Hayao Taguchi, Masahiro Nakajima, Shinya Fushinobu
- DOI番号
- :10.1074/jbc.RA117.001536
- 論文URL
- :http://www.jbc.org/content/early/2018/04/20/jbc.RA117.001536.short
問い合わせ先
教授 伏信 進矢(ふしのぶ しんや)
Tel:03-5841-5151
Fax:03-5841-5151
研究室URL:http://enzyme13.bt.a.u-tokyo.ac.jp/
用語解説
- 注1 バイオセンサー
- 生体分子、生物体の高い選択性を利用し、特定の物質を感知するように設計されたセンサー。
- 注2 β-1,2-グリコシド結合
- 澱粉はグルコースのC1位のα-アノマーとC4位が繋がったα-1,4-グリコシド結合、セルロースはC1位のβ-アノマーとC4位が繋がったβ-1,4-グリコシド結合からなるが、β-1,2-グルカンはC1位のβ-アノマーとC2位が繋がったβ-1,2-グリコシド結合からなる。糖のC1位とC2位は位置が近く、分子内の干渉が生じやすいため、この位置で繋がった多糖は希少な場合が多い。
- 注3 溶質結合タンパク質
- 細胞の表面で特定の物質と強く結合し、ABC輸送体による細胞膜の通過を仲介するタンパク質。
- 注4 等温滴定熱量測定(Isothermal Titration Calorimetry, ITC)
- 溶液中の分子間の相互作用に伴う微小な熱量変化を直接測定する手法であり、生体分子間の相互作用を調べるのに広く用いられる。相互作用の強さ(結合定数)に加え、結合比や、結合にともなうエンタルピー変化、エントロピー変化などの熱力学的な性質を表す数値を算出することができる。
- 注5 X線結晶構造解析
- タンパク質の立体構造を決定するための手法の一つ。目的タンパク質の結晶にX線を照射し、回折データを取得することにより、タンパク質内の電子密度の情報が得られ、その微細な三次元構造を知ることができる。
- 注6 分子動力学シミュレーション
- 分子集団を構成する各原子に対し、経験的なポテンシャル(力場)の下、運動方程式を逐次的に解くことにより全原子の軌跡を求めるコンピューターシミュレーションの手法。タンパク質の動的な状態を調べるのに広く用いられており、得られた軌跡から自由エネルギーをはじめとする様々な物理量を計算することができる。
- 注7 部位特異的変異導入
- 人工的に遺伝子に変異を導入することにより、そこから作られるタンパク質を構成する特定の部分(アミノ酸残基)を改変する手法。タンパク質の機能に関わる部位を特定したり、タンパク質の機能を改変するために用いられる。