キクタニギクのゲノムを解読、開花に関わる遺伝子探索へ 〜栽培ギクの起源を明らかにし、品種改良を加速〜
- 発表者
-
平川 英樹(かずさDNA研究所 ゲノム情報解析施設 施設長)
住友 克彦(農業・食品産業技術総合研究機構 野菜花き研究部門 花き遺伝育種研究領域 上級研究員)
久松 完(農業・食品産業技術総合研究機構 野菜花き研究部門 花き生産流通研究領域 上級研究員)
永野 聡一郎(かずさDNA研究所 植物ゲノム・遺伝学研究室:研究当時)
白澤 健太(かずさDNA研究所 植物ゲノム・遺伝学研究室 主任研究員)
樋口 洋平(東京大学大学院農学生命科学研究科 生産・環境生物学専攻 講師)
草場 信(広島大学大学院理学研究科 教授)
腰岡 政二(日本大学 生物資源科学部 生命農学科 教授)
中野 善公(農業・食品産業技術総合研究機構 野菜花き研究部門 花き生産流通研究領域 主任研究員)
八木 雅史(農業・食品産業技術総合研究機構 野菜花き研究部門 花き遺伝育種研究領域 上級研究員)
山口 博康(農業・食品産業技術総合研究機構 野菜花き研究部門 花き遺伝育種研究領域 ユニット長)
谷口 研至(広島大学大学院理学研究科 特任准教授)
中野 道治(広島大学大学院理学研究科 特任助教)
磯部 祥子(かずさDNA研究所 植物ゲノム・遺伝学研究室 室長)
発表のポイント
- キクタニギクのゲノム解析を行い、全ゲノムの89%にあたる2.72Gbの配列を解読しました。 解読した配列から推定された遺伝子数は71,057です。開花に関わる遺伝子の探索と栽培ギクのゲノム配列変異の検出などを行いました。
- キクタニギクはキク科キク属の植物で、日本では東北から九州にかけて自生しています。栽培ギクに比べてゲノム構造が単純なため、キクのモデル植物としてさまざまな研究に利用されています。
- キクタニギクのゲノムや開花に関わる遺伝子が明らかになったことで、キクの開花制御や花の形態形成に関する研究を一層すすめることができます。そして、これらの情報を利用した栽培ギクの育種の効率化が進むと期待できます。
発表内容
写真:キクタニギク(広島大学提供)

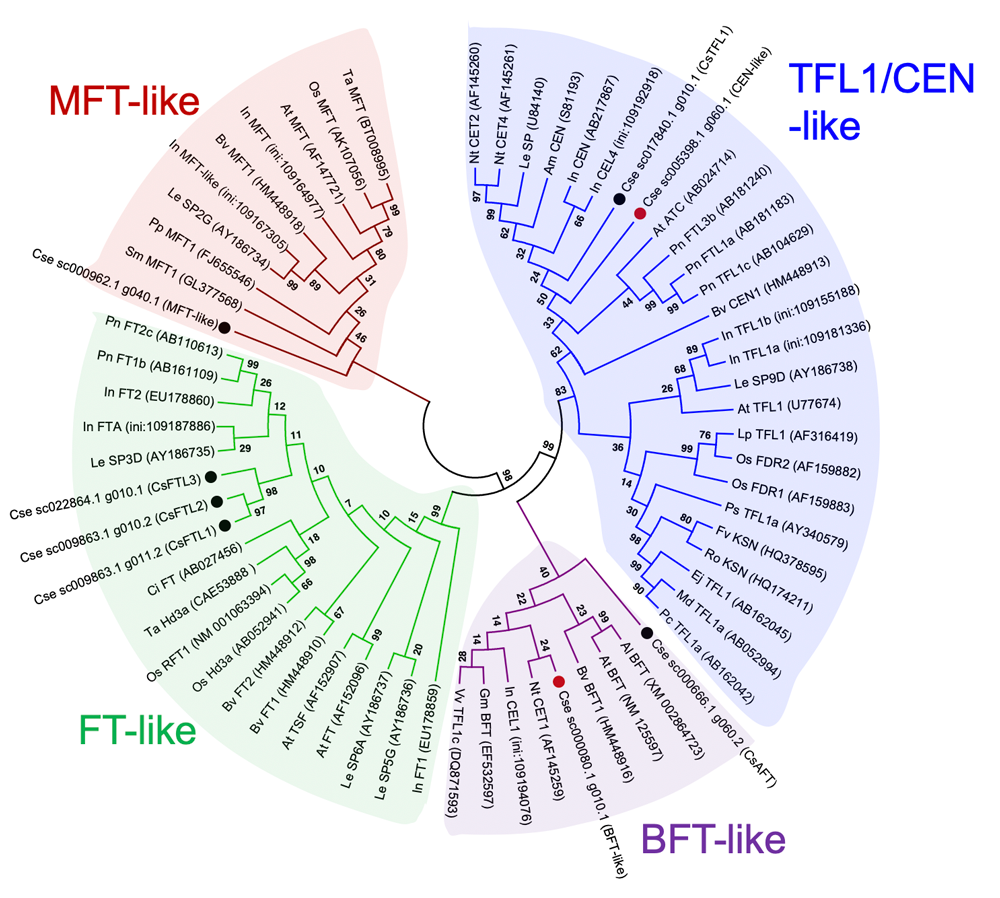
図1 探索した開花に関連する遺伝子のうち、花成ホルモン(フロリゲン)を含むPEBP (phosphatidylethanolamine-binding protein) ファミリーに属するに関連する遺伝子群と他種で同定されている遺伝子との配列の類似性を示した系統樹。黒丸は既報のキクタニギク遺伝子。赤丸はゲノム解読により初めて推定された遺伝子。

図2 キクタニギクの自殖可能な系統「AEV2」と他殖系統「NIFS-0」のゲノム配列を比較し、検出された変異を染色体に沿ってならべた連鎖地図。AeG1~AeG9が「AEV2」の連鎖地図、NiG1~NiG9が「NIFS-0」の連鎖地図。赤色は変異が多く検出された部位。グレーは対となる染色体のうち片側の染色体に偏って後代に遺伝した部位。自殖可能な系統「AEV2」ではAe5とAe6で偏りが顕著だった。
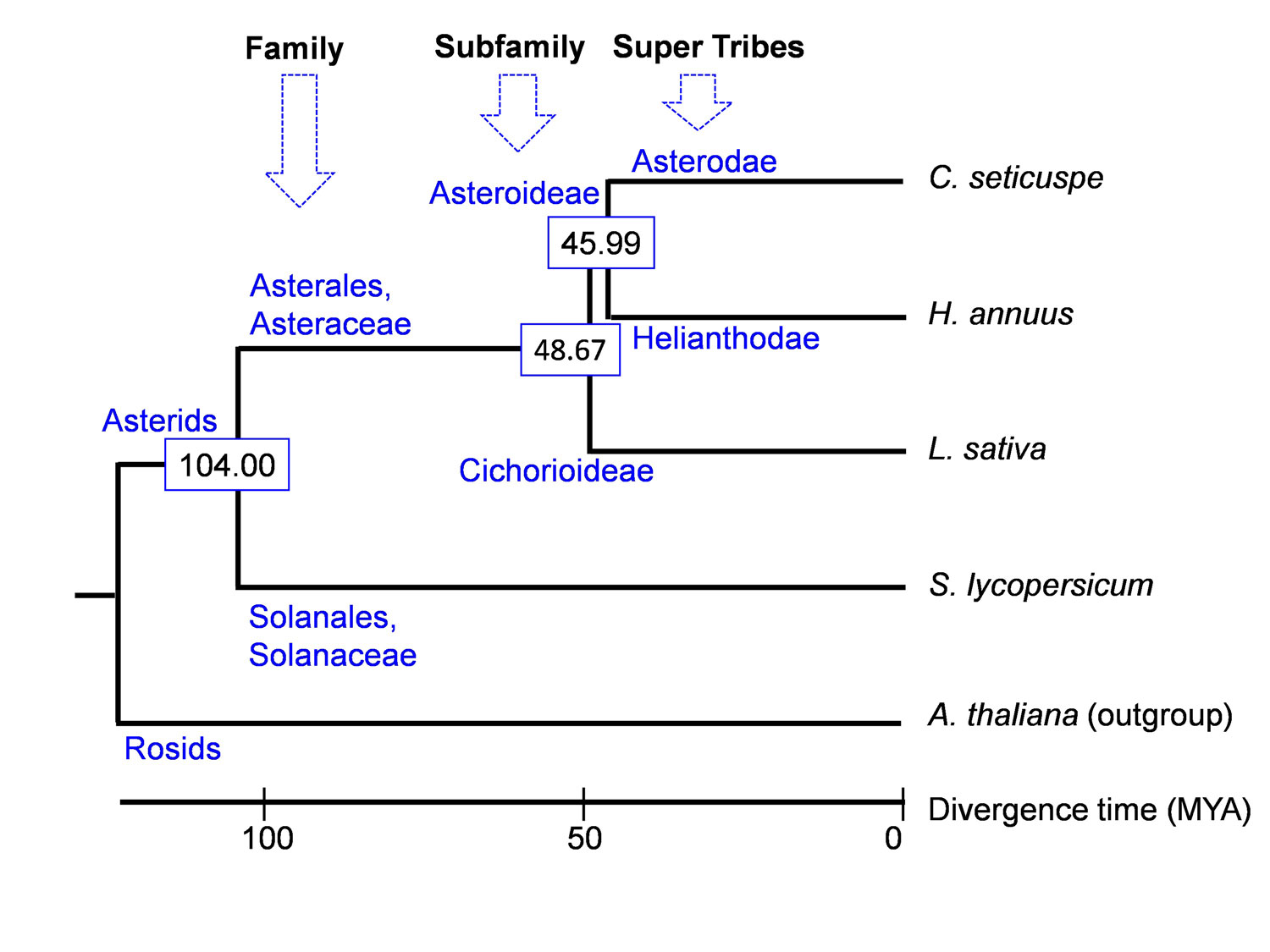
図3 キクタニギク(C. seticuspe:キク科キク属)とヒマワリ(H.annus:キク科ヒマワリ属)、レタス(L. sativa:キク科アキノノゲシ属)、トマト(S. lycopersicum:ナス科ナス属)、シロイヌナズナ(A. thaliana:アブラナ科シロイヌナズナ属)の分岐の推定(図内の数字は推定された分岐年(百万年単位))。およそ4600万年前にキクタニギクとヒマワリが分岐したと推定される。
【背景】
キク科に属する植物種は被子植物の約10%を占め、特に栽培ギクの起源となったキク属は主に東アジアで分化したことから、植物の多様性を研究する上で重要な植物群のひとつです。また、栽培ギクは日本の切花生産の約40%を占めており、世界的にもバラやカーネーションと並んで重要な花き品目です。今回解析したキクタニギク(Chrysanthemum seticuspe)は、キク科キク属の多年草で、東北から九州にかけて自生しており、10~11月に黄色い花をつけます。キクタニギクの染色体数は2n=18ですが、栽培されているキクは多くの野生ギクが交雑を繰り返し品種として成立したため、複雑なゲノム構造になっています。そのため、ゲノム構造が単純なキクタニギクが、キクのモデル植物として開花制御の研究などに利用されています。
キク属の遺伝学的研究がこれまで進んでいなかった理由としては、自家不和合性もあります。通常のキクタニギクは、同じ個体の花にある雄蕊(ゆうずい;おしべ)の花粉が雌蕊(しずい;めしべ)についても種子がほとんどできません(他殖性、と言います)。そのため、遺伝学的な研究に重要な、ゲノム構造が同質で安定した形質が現れる、いわゆる「純系」と呼ばれる系統をつくることができなかったのです。広島大学は、同一個体の花粉で種子を作ることができる(自殖性)個体「AEV2」を発見し、文部科学省・ナショナルバイオリースプログラムのもと、この「AEV2」を用いて5回の自殖を繰り返しました。そして、ゲノム配列の構造がより同質化された系統「XMRS10」を作出し、ゲノム解析を行いました。
本研究では、広島大学で作出された「XMRS10」を用いて、かずさDNA研究所および農研機構野菜花き研究部門でゲノム配列の解読と遺伝子予測、連鎖地図の作成を行いました。また、農研機構野菜花き研究部門と日本大学、および東京大学大学院農学生命科学研究科の樋口講師らは、生育・開花に関わる遺伝子の探索を実施しました。キクタニギクのゲノムサイズの推定は農研機構野菜花き研究部門で、栽培キクゲノムの配列変異の検出はかずさDNA研究所で行いました。
キクタニギクは、文部科学省・ナショナルバイオリソースプロジェクト(NBRP)広義キク属で、バイオリソースとして扱われています。プロジェクトについては、HPをご覧ください。
NBRP広義キク属:http://shigen.nig.ac.jp/chrysanthemum/index.jsp
【研究成果の概要と意義】
- 超並列シークエンシング技術と大型計算機を駆使し、キクタニギクの全ゲノムの89%にあたる27億2000万塩基対(2.72Gb)の配列を解読しました。
- 解読された配列から推定された遺伝子数は71,057でした。このうち開花に関わる遺伝子として221の遺伝子がみいだされました。
- ゲノム配列から開花関連の遺伝子を探索し、シロイヌナズナなどで花成抑制遺伝子として報告されているCEN様遺伝子をあらたに見つけることができました。また、日長や温度処理を変えたときのキクタニギク茎頂での遺伝子発現解析により、新たに見つかった遺伝子は季節変動に応じて開花・休眠を制御する遺伝子である可能性が示唆されました。
- 解読したゲノム配列を用いて、自殖系統と他殖系統の個体群がそれぞれもつ、配列の変異を染色体上に並べた連鎖地図を作成しました。
- 解読したゲノムと栽培ギク6系統のゲノム配列を比較して、合計で95万4706の配列の違い(1塩基多型、SNPs)を検出しました。このうち533の変異は、遺伝子機能の変異に関わる可能性が極めて高い変異であると推定されました。
- キクタニギクで推定された遺伝子配列を、同じキク科のヒマワリ、レタスの遺伝子配列と比較したところ、キクタニギクはヒマワリとより近縁であり、およそ4600万年前にキクタニギクとヒマワリが分岐したと推定できました。
- 本研究により得られた情報は、データベースとして公開しています。
MumGARDEN (http://mum-garden.kazusa.or.jp/)
【将来の波及効果】
- 世界で初めてキクタニギクのゲノム解読に成功したことで、栽培キクのモデル植物であるキクタニギクや栽培ギクの遺伝子の解明などの研究を進めることができます。
- 栽培ギクではこれまでゲノム情報を用いた育種があまり実施されていませんでしたが、キクタニギクのゲノム配列情報を用いることで、栽培ギクの有用形質を選抜するためのゲノム情報を得やすくなり、栽培ギクの育種が進むことが期待できます。
- キクは古くから電照により開花を制御するなど、開花日を調節した栽培が実施されてきました。キクタニギクで探索された開花に関する遺伝子配列の機能解析をすすめて、キクの開花メカニズムを明らかにすることで、より精度の高いキクの栽培法の開発につながることが期待できます。
発表雑誌
- 雑誌名
- DNA Research
- 論文タイトル
- De novo whole-genome assembly in Chrysanthemum seticuspe, a model species of Chrysanthemums, and its application to genetic and gene discovery analysis.
- 著者
- Hideki Hirakawa†, Katsuhiko Sumitomo†, Tamotsu Hisamatsu, Soichiro Nagano, Kenta Shirasawa, Yohei Higuchi, Makoto Kusaba, Masaji Koshioka, Yoshihiro Nakano, Masafumi Yagi, Hiroyasu Yamaguchi, Kenji Taniguchi, Michiharu Nakano and Sachiko N Isobe*.(†These authors contributed equally to this work, *corresponding author)
- DOI番号
- 10.1093/dnares/dsy048
- 論文URL
- https://academic.oup.com/dnaresearch/advance-article/doi/10.1093/dnares/dsy048/5303208
(本研究成果はDNA Research誌で1月27日にオンライン公開されました。)
問い合わせ先
公益財団法人かずさDNA研究所
植物ゲノム・遺伝学研究室 室長 磯部 祥子(いそべ さちこ)
TEL:0438-52-3930
E-mail:sisobe<アット>kazusa.or.jp <アット>を@に変えてください。
東京大学大学院農学生命科学研究科 生産・環境生物学専攻 園芸学研究室
講師 樋口 洋平(ひぐち ようへい)
Tel:03-5841-1131
E-mail:ahigu<アット>mail.ecc.u-tokyo.ac.jp <アット>を@に変えてください。
用語解説
- 注1 遺伝子
親から子へと遺伝する、あるいは細胞から細胞へと伝えられる形質を決定する因子であり、生物の体を作り動かすのに必要なタンパク質などを作るための設計図のことで、その本体はDNAである。 - 注2 ゲノム
生物をその生物たらしめるのに必須な最小限の染色体のひとまとまり、またはDNA全体のことをいう。

