コレステロール代謝を制御する転写因子SREBPの新たな活性化機構の発見
- 発表者
- 官 彦州(東京大学大学院農学生命科学研究科・学振外国人特別研究員)
高橋 裕(東京大学大学院農学生命科学研究科・助教)
丸山 貴史(東京大学大学院農学生命科学研究科・博士課程)
清水 誠(東京大学大学院農学生命科学研究科・特任准教授)
山内 祥生(東京大学大学院農学生命科学研究科・准教授)
佐藤隆一郎(東京大学大学院農学生命科学研究科・教授)
発表のポイント
- コレステロール・脂肪酸代謝を制御する転写因子SREBP(注1)の活性化の新たな機構を発見しました。
- SREBPと複合体を形成するSCAP(注2)にユビキチン化酵素RNF5(注3)が結合し、ポリユビキチン化修飾を行い、SREBPの活性化を促すことがわかりました。
- 新たな調節機構の発見により脂質代謝制御機構の詳細が明らかになり、SREBP活性を減弱させて代謝改善を目指す、治療薬・機能性食品開発の新たな展開が期待されます。
発表概要
細胞内のコレステロール量は厳密に制御されています。細胞外からLDLを取り込みそこに含まれるコレステロールとともに、細胞自身がコレステロールを合成し細胞内のコレステロール量は増加します。このような状況下で、転写因子SREBPの活性化は抑制され、コレステロール合成・取り込みに関与する遺伝子群の発現は負に制御され、フィードバック機構が成立します。種々の代謝疾患においてSREBPの過剰な活性化が発症の引き金になっていることから、SREBP活性化機構の理解を深め、これを抑制する方法を解明することが求められています。
今回、東京大学大学院農学生命科学研究科の佐藤隆一郎教授のグループは、SCAPをポリユビキチン化修飾する酵素としてRNF5を見出し、この修飾がSREBP-SCAP複合体の小胞体からゴルジへの輸送を促進し、SREBPを活性化するという新たな調節機構を発見しました。この機構を抑制する薬物・機能性食品成分には脂質代謝異常を改善する効果が期待されます。
発表内容
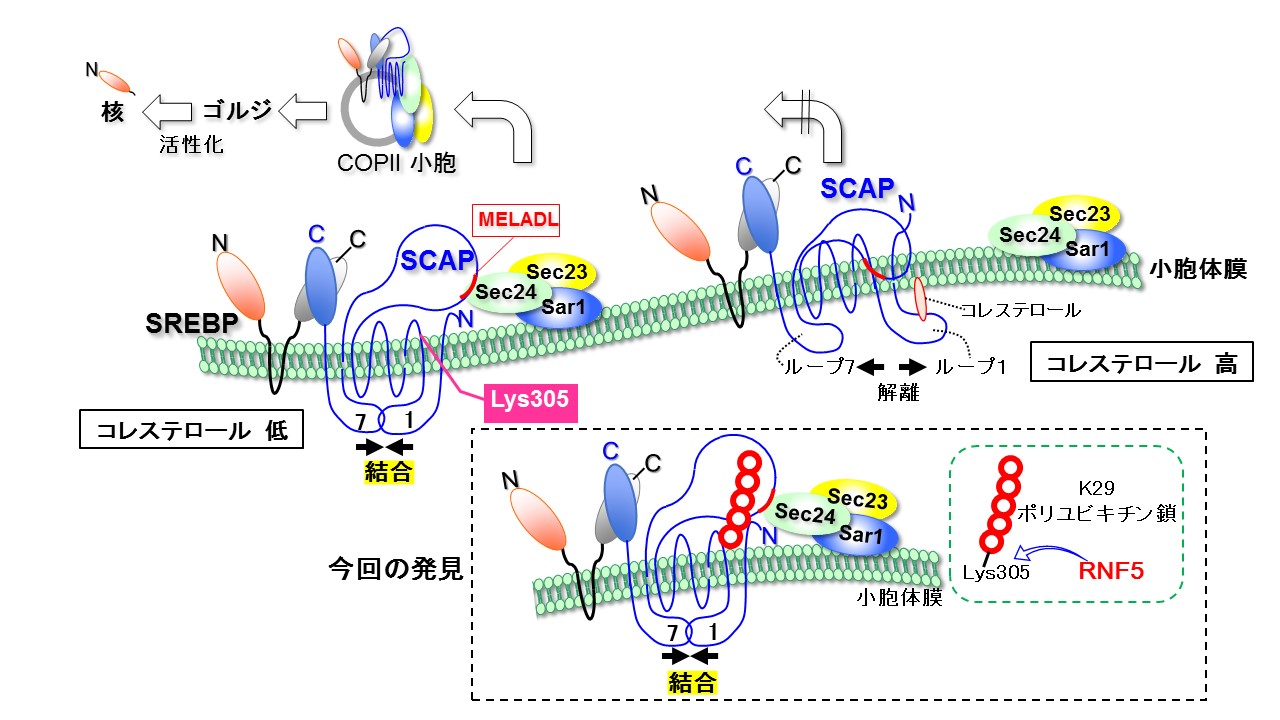
図1 小胞体膜上におけるSREBP/SCAP複合体のゴルジ輸送の調節機構モデル
細胞内コレステロール量が低い時、SCAP分子の小胞体内腔側のループ1とループ7が結合し、このことが細胞質側のループの構造変化をもたらし、MELADL配列(赤線)が露出します。Sec24がこの配列に結合し、この複合体はCOPII小胞へと積み込まれ、ゴルジ装置へと輸送されます。ゴルジ装置では切断酵素がSREBPのN末端領域を切り出し、この活性型が核へと輸送され、核内で転写因子としてコレステロール代謝関連遺伝子の発現を正に制御します。コレステロール量が多い時には、ループ1と7の結合は解除され、SCAPとSec24が結合することはありません。その結果として、コレステロール代謝関連遺伝子の発現は低下します。今回の研究で、SCAPの305番目のLys残基がRNF5によりポリユビキチン化修飾を受けることが明らかになりました。このポリユビキチン修飾はユビキチン分子の29番目のLys残基(K29)を介した報告例の少ないタイプで、ループ1と7の結合を促し、SREBPの活性化を亢進させる働きを持つことが明らかになりました。
転写因子SREBPは小胞体膜上で結合タンパク質SCAPと2量体を形成します(図1)。この複合体は細胞内のコレステロール量が低値の時にはゴルジ装置まで輸送され、2種類の切断酵素によるプロセシングを受けた後に、N末端側の活性型SREBPが核へと移行し、核内で転写因子として種々の応答遺伝子の転写をスイッチオンします。SCAPは8回膜貫通領域を持つ膜タンパク質で、SREBPと複合体を形成し、小胞体からゴルジへの輸送をコントロールしています。細胞内のコレステロール量が低い時には、SCAPの小胞体内腔側に突き出したループ1とループ7が結合し(図参照)、その結果構造変化がもたらされ、SCAPの細胞質側のループのMELADL(Met-Glu-Leu-Ala-Asp-Leu)配列が露出され、そこにCOPII小胞上のタンパク質Sec24が結合します。こうしてSREBP-SCAP複合体はSec23/24-Sar1複合体に引きずられるようにして、COPII小胞へと乗り込み、ゴルジまで輸送されます。一方、コレステロール量が増えると、ループ1にコレステロールが結合することが知られており、その結果、ループ1とループ7の結合は解除され、MELADL配列が露出されなくなります。こうしてSREBP-SCAP複合体は小胞体膜上に留まり、核の中で転写因子として働くことはできません。
今回我々は、SCAPをユビキチン修飾する酵素(E3 ligaseと呼ばれる)RNF5を見出しました。SCAPは比較的早い分解を受けるタンパク質であることを我々は前回の論文(Kuan et al. J. Biol. Chem. 292, 3106-3028, 2017)で示しています。RNF5によるユビキチン化はSCAPの分解を促進するものと予想しましたが、実際には分解を促すことはありませんでした。予想外なことに、RNF5を過剰発現させるとSREBPの活性化が亢進し、siRNAを用いて発現を抑制するとSREBPの活性化は減弱しました。ユビキチン修飾は通常、基質のLys残基に生じます。SCAPには18個のLys残基が存在しますが、それらをArg残基に置換したSCAPを作成し、RNF5によるユビキチン化を解析しました。その結果、305番目のLys残基がRNF5による特異的なユビキチン化を受けることが明らかになりました。それ以外のLys残基もRNF5以外のユビキチン化酵素E3 ligaseにより修飾されていることもわかりました。ユビキチン分子は76個のアミノ酸からなるポリペプチドであり、その分子内には7個のLys残基が存在します。48番目のLys残基に別のユビキチン分子のC末端のGly残基がアミド結合して鎖が伸長すると、プロテアソームによる分解へと導かれることが知られています。そこでユビキチン分子中のLys残基をArg残基へと置換した様々な変異体を作成し、RNF5によるSCAPのユビキチン化を解析したところ、29番目のLys残基(K29)を介して、ポリユビキチン鎖が伸長されることが明らかになりました。K29を介したポリユビキチン化修飾の例はこれまでに数少なく、タンパク質-タンパク質結合に関与するという報告があります。特殊な解析系を構築して、RNF5によりSCAPのユビキチン化を亢進させると、ループ1とループ7の結合が増加することを確認しました。SCAPの305番目のLys残基をArg残基に置換しておくと、ループ間の結合は増加しませんでした。つまり、SCAPの305番目のLys残基へのK29を介したポリユビキチン修飾はSCAPの構造変化をもたらし、その結果、コレステロールが不足した状況を模倣することがわかりました。
上述したように種々の代謝疾患においてSREBPの過剰な活性化が発症の引き金になっていることから、SCAPの305番目のLys残基を介したポリユビキチン化を抑制することは、活性化の減弱に繋がることが期待されます。RNF5は他の基質のユビキチン化にも関与しますので、その酵素活性を直接阻害するのは賢明とは言えません。SCAPの305番目のLys残基へのユビキチン化を抑制する薬物・機能性食品成分はSREBP活性化を負に制御することが可能です。創薬・機能性食品開発の新たな標的として、今後の研究の進展が期待されます。
この研究は、日本学術振興会科研費基盤(S)、AMED-CRESTの支援を受けて行われました。
発表雑誌
- 雑誌名
- Journal of Biological Chemistry
- 論文タイトル
- Ring finger protein 5 activates sterol regulatory element-binding protein 2 (SREBP2) to promote cholesterol biosynthesis via ubiquitinating SREBP chaperone SCAP.
- 著者
- Yen-Chou Kuan, Yu Takahashi, Takashi Maruyama, Makoto Shimizu, Yoshio Yamauchi and Ryuichiro Sato
- DOI番号
- 10.1074/jbc.RA119.011849
- 論文URL
- https://www.jbc.org/content/early/2020/02/13/jbc.RA119.011849.abstract
問い合わせ先
東京大学大学院農学生命科学研究科 応用生命化学専攻 食品生化学/栄養・生命科学研究室
教授 佐藤 隆一郎(さとう りゅういちろう)
Tel:03-5841-5136
Fax:03-5841-8029
E-mail:aroysato<アット>mail.ecc.u-tokyo.ac.jp <アット>を@に変えてください。
用語解説
- 注1 SREBP
コレステロール代謝関連遺伝子の発現を主に制御するSREBP-2と脂肪酸代謝関連遺伝子の発現を調節するSREBP-1が存在する。いずれも2回の膜貫通領域を持ち、合成後、小胞体膜上に膜タンパク質として局在する。 - 注2 SCAP
8回膜貫通領域を持つSREBP結合タンパク質。SREBPのC末端領域とSCAPのC末端領域が結合し、2量体を小胞体膜上で形成する。細胞内のコレステロール量が低下した条件などに呼応して複合体はゴルジ装置へと輸送され、そこで切断を受け、SREBPのN末端領域が活性型転写因子として核へ移行する。 - 注3 RNF5
小胞体膜上に局在し、基質のLys残基にユビキチン分子をアミド結合させる酵素(E3 ligase)。複数の基質をポリユビキチン鎖で修飾して、これらを分解へと誘導することが報告されている。

