ゲノム系統地理学が明らかにした交雑によるミトコンドリアゲノムの進化
- 発表者
- 平瀬 祥太朗 (東京大学大学院農学生命科学研究科 附属水産実験所 助教)
手塚 あゆみ (龍谷大学 農学部:当時)
永野 惇 (龍谷大学 農学部)
佐藤 茉菜 (東京大学大学院農学生命科学研究科 附属水産実験所 特任研究員)
細谷 将 (東京大学大学院農学生命科学研究科 附属水産実験所 助教)
菊池 潔(東京大学大学院農学生命科学研究科 附属水産実験所 教授)
岩崎 渉(東京大学大学院理学系研究科 生物科学専攻 准教授)
発表のポイント
- 「ミトコンドリアゲノムと核ゲノム間の不適合(ミト-核ゲノム不適合)」が、遺伝的に分化した生物集団間における交雑によって生じた可能性を示しました。
- この不適合は、ミトコンドリアゲノムに進化をもたらすと考えられます。
- “ゲノム系統地理学”(注1)によって、生物の進化をゲノム・時間・空間から総合的に解明できます。
発表概要
遺伝的に異なる集団間の交雑で生じたゲノム変異の新たな組み合わせは、遺伝子間の協調を妨げ、交雑個体の生存能力や妊性を低下させると考えられています。このような現象はゲノム不適合と呼ばれ、新たな遺伝子の組み合わせを持つ生物の誕生を抑制することから、生物種の進化を考える上で重要な要素です。特に、ミトコンドリアにおけるエネルギー生産活動に関与する複数の遺伝子は、ミトコンドリアが有するミトコンドリアゲノムと細胞核が有する核ゲノムのそれぞれに存在していますが、ミトゲノムと核ゲノム間の不適合(ミト-核ゲノム不適合)がそうした交雑によって生じることが予測されています。本研究では、ハゼ科の魚であるアゴハゼの2つの種内系統の交雑によってミト-核ゲノム不適合が生じ、それを解消するためにミトゲノムの進化が起きた可能性を全ゲノムレベルでの遺伝解析で明らかにしました。
発表内容
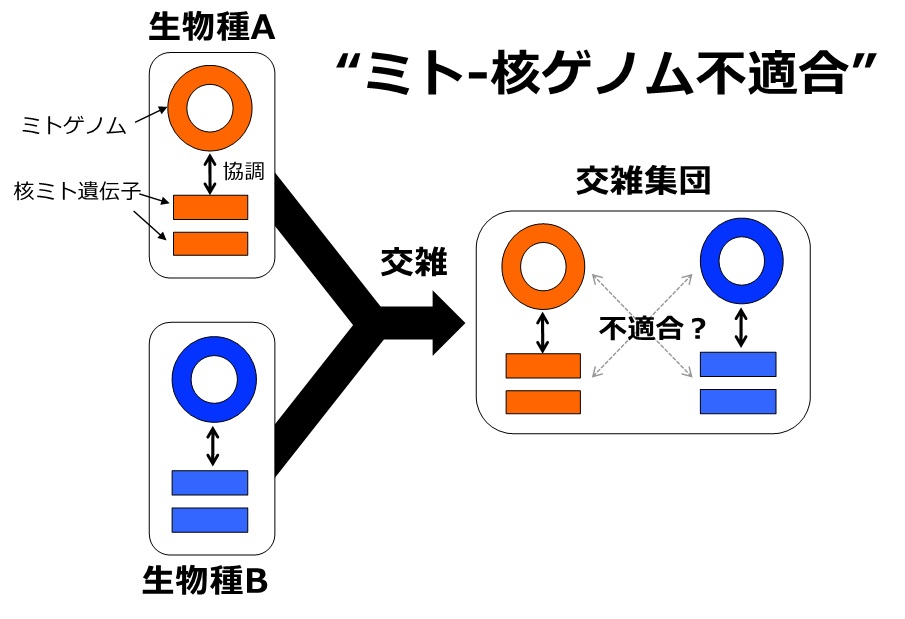
図1 ミト-核ゲノム不適合のイメージ図
生物種Aと生物種Bのそれぞれにおいて、ミトゲノムと核ミト遺伝子間は協調して働くことができるよう独自に進化している。しかし、交雑が起こると、生物種Aのミトゲノムと生物種Bの核ミト遺伝子、生物種Bのミトゲノムと生物種Aの核ミト遺伝子といった、これまでになかった組み合わせができ、ミト-核ゲノム不適合が生じる。
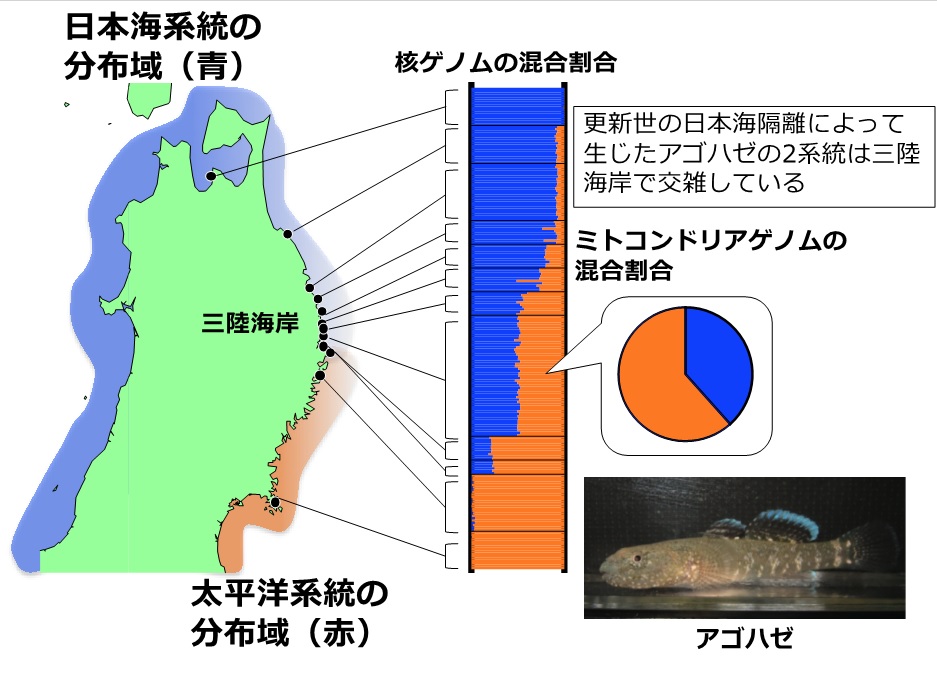
図2 三陸海岸周辺におけるアゴハゼの太平洋系統と日本海系統の交雑
日本海系統は、津軽海峡越えて三陸海岸まで分布を広げており、そこで太平洋系統との交雑が生じている。三陸海岸の北部から南部にかけて2つの系統の核ゲノムが少しずつ混ざっており、その中心に存在する交雑集団では2つの系統のミトゲノムと核ゲノムとが混在している。
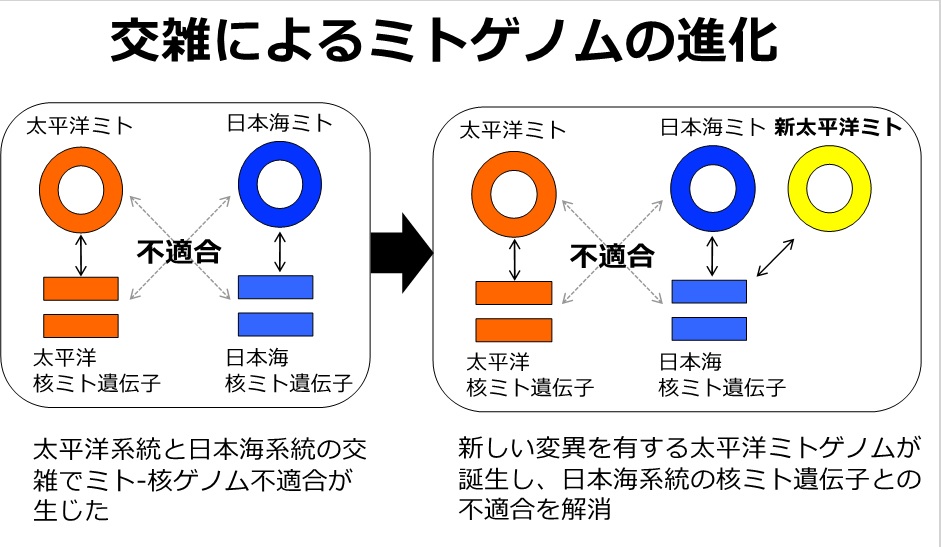
図3 ゲノム系統地理によって示された交雑によるミトゲノム進化のイメージ図
ゲノムは、あらゆる生物の生命活動を支える最も重要な基盤であり、それぞれの生物種において親から子へ、子から孫へと受け継がれながら、独自の変異が蓄積されていきます。こうして独自に進化してきた生物同士が交雑すると、これまでになかったゲノム変異の組み合わせ持つ個体が生まれ、その個体では遺伝子間における正常な相互作用が妨げられることで、生存能力や妊性を低下すると考えられています。このような現象は「ゲノム不適合」と呼ばれています。
ミトコンドリア内に存在するミトコンドリアゲノム(ミトゲノム)と、細胞核内の核ゲノムに存在するミトコンドリア関連遺伝子(核ミト遺伝子)は、こうした不適合が生じる可能性が高い遺伝子群の1つです(ミト-核ゲノム不適合;図1)。ミトコンドリアは、人類をはじめとした真核生物が呼吸によってエネルギーを生産するための“工場”ですが、その活動において、ミトゲノムと核ミト遺伝子間の協調は不可欠であり、なおかつ、それらの遺伝子は協調して働くことができるよう生物ごとに独自に進化しているからです。実際に、異なる生物由来の核ゲノムとミトゲノムを持つ個体の生存能力が下がることが、実験室での交配実験やサイブリッド(注2)によって示唆されています。一方、野外の生物集団を対象とした分子系統地理学(注3)的研究は、遺伝的に分化した生物集団間の交雑によって、核ゲノムとミトゲノムの新たな組み合わせを持つ集団が形成されている例を数多く報告しています。したがって、このミト-核ゲノム不適合が自然界において生じているかどうかについては未だ議論が絶えません。
仮に、ミト-核ゲノム不適合が実際に引き起こされるとして、異なる生物集団由来のミトゲノムと核ミト遺伝子が野外で生じた交雑によって一つの集団内で完全に混ざってしまったらどうなるでしょう。この場合には、ミト-核ゲノム不適合を起こさない遺伝子の組み合わせや変異を持つ個体が自然選択(注4)によって多くなり、交雑集団のゲノム構成が少しずつ変化していくと予想されます。したがって、そのような交雑集団のゲノム構成を徹底的に調査すれば、自然界におけるミト-核ゲノム不適合の痕跡を見つけることができるかもしれません。
本研究では、三陸海岸のアゴハゼ(Chaenogobius annularis)に着目しています。アゴハゼは日本列島と朝鮮半島沿岸の岩礁性海岸に分布するハゼ科の魚で、潮の干満によって形成される潮だまり(タイドプール)で観察することができ、子供たちの磯遊びでは恰好の遊び相手になっています。これまでの分子系統地理学研究により、アゴハゼの種内には更新世氷期の日本海隔離(注5)によって遺伝的に分化した太平洋系統と日本海系統が存在し、この2つの系統が交雑して誕生した集団が、三陸海岸で長い間存続していることがわかっていました。しかも、この交雑集団では、2つの系統のミトゲノムと核ゲノムとが完全に混ざっているのです(図2)。
そこで、この交雑集団のゲノム構成を、MiSeqやHiSeq、PacBioなどのシーケンサーを用いたde novo ゲノムアセンブリ(注6)、全ゲノムリシーケンス(注7)、RNAシーケンス(注8)、RADシーケンス(注9)、ミトゲノムシーケンスによって徹底的に調査しました。その結果、この交雑集団が有する太平洋系統のミトゲノム(太平洋ミトゲノム)のいくつかの遺伝子において、アミノ酸を変化させ、機能に影響を与える可能性のある変異が生じており、自然選択が働いた可能性が示されました。さらに、これらの変異によって太平洋ミトゲノムの遺伝子の2つのアミノ酸が、日本海系統のミトゲノムのアミノ酸配列に変化していることがわかりました。また、この新しい太平洋ミトゲノム(新太平洋ミトゲノム)は、日本海系統の核ミト遺伝子と一緒に個体に受け継がれている一方、従来の太平洋ミトゲノムは太平洋系統の核ミト遺伝子と受け継がれていました。以上の結果から、太平洋ミトゲノムと日本海系統の核ミト遺伝子との不適合が生じ、その不適合を解消するような新太平洋ミトゲノムが、交雑集団に生まれたと結論づけられました(図3)。
分子系統地理学は、生物集団が長い歴史の中で、どのように分岐・拡大してきたかを解明してきました。本研究のような分子系統地理学とゲノミクスとの融合は、自然界で生じてきた生物集団の進化をゲノム・時間・空間という視点から総合的に解明していくことにつながります。
発表雑誌
- 雑誌名
- Evolution
- 論文タイトル
- Integrative genomic phylogeography reveals signs of mitonuclear incompatibility in a natural hybrid goby population
- 著者
- Shotaro Hirase*, Ayumi Tezuka, Atsushi J. Nagano, Mana Sato, Sho Hosoya, Kiyoshi Kikuchi, Wataru Iwasaki*
- DOI番号
- 10.1111/evo.14120
- 論文URL
- https://onlinelibrary.wiley.com/doi/epdf/10.1111/evo.14120
問い合わせ先
東京大学大学院農学生命科学研究科 附属水産実験所
助教 平瀬 祥太朗(ひらせ しょうたろう)
Tel:053-592-2821
Fax:053-592-2822
E-mail:cashirase<アット>g.ecc.u-tokyo.ac.jp <アット>を@に変えてください。
用語解説
- 注1 ゲノム系統地理学
生物が歴史の中でどのように分岐・拡大してきたかを解明する分子系統地理学が、ゲノミクスと融合した研究。 - 注2 サイブリッド
ミトコンドリアゲノムを持たない細胞と、核をもたない細胞を融合させて作製した細胞。ミトコンドリアのみが異なる細胞をつくり出すことによって、ミトコンドリアゲノムが細胞の機能に与える影響を調べることができる。 - 注3 分子系統地理学
近縁な集団間の空間的・時間的な関係性を、DNA情報を用いて明らかにし、それらの分布域形成の歴史を推定する学問分野。例えば、同一の種であっても、地域集団間で遺伝的分化が生じていた場合には、それらの地域間には過去に何らかの地理的障壁があり、移動分散が制限されていた可能性がある。 - 注4 自然選択
生存に有利な変異を持つ個体が生き残ること。 - 注5 日本海隔離
日本海は浅い海峡によって周辺の海域とつながっているが、更新世の氷期には海水準が低下し海峡が陸化したことで、日本海は周辺の海域から隔離されていた。この日本海の隔離が、複数の沿岸性生物の太平洋側と日本海側の集団間の遺伝的分化を引き起こしたと考えられている。 - 注6 de novo ゲノムアセンブリ
生物種の全ゲノムの塩基配列を新規に構築し、変異の検出などを行う上で基準となる“参照ゲノム”を得ること。 - 注7 全ゲノムリシーケンス
参照ゲノムが構築された生物種の全ゲノム配列を再度決定すること。参照ゲノムと比較することで、変異を検出することができる。 - 注8 RNAシーケンス
生物種の全転写産物の塩基配列を決定すること。 - 注9 RADシーケンス
Restriction Site Associated DNA(RAD)シーケンス。制限酵素認識サイトの近隣のゲノム領域の塩基配列を決定し、参照ゲノムと比較することで変異を検出する。

