リンゴの品種改良に貢献した起源品種の遺伝領域〜起源品種のハプロタイプの遺伝を自動的に追跡する方法の開発〜
- 発表者
- 南川 舞 (東京大学 大学院農学生命科学研究科 生産・環境生物学専攻 特任研究員:当時/
現 日本学術振興会 特別研究員)
國久 美由紀 (農研機構 果樹茶業研究部門 品種育成研究領域 上級研究員)
野下 浩司 (九州大学 大学院理学研究院・植物フロンティア研究センター 助教)
森谷 茂樹 (農研機構 果樹茶業研究部門 リンゴ研究領域 主任研究員)
阿部 和幸 (農研機構 果樹茶業研究部門 品種育成研究領域 領域長)
林 武司 (農研機構 次世代作物開発研究センター 基盤研究領域 ユニット長:当時)
片寄 裕一 (国立研究開発法人農業生物資源研究所 農業生物先端ゲノム研究センター 先端ゲノム解析室:当時/
現 農研機構 次世代作物開発研究センター ゲノム育種研究統括監)
松本 敏美 (国立研究開発法人農業生物資源研究所 農業生物先端ゲノム研究センター 先端ゲノム解析室:当時/
現 農研機構 生物機能利用研究部門 動物機能利用研究領域 契約研究員)
西谷 千佳子 (農研機構 果樹茶業研究部門 品種育成研究領域 上級研究員)
寺上 伸吾 (農研機構 果樹茶業研究部門 品種育成研究領域 主任研究員)
山本 俊哉 (農研機構 果樹茶業研究部門 品種育成研究領域 ユニット長:当時)
岩田 洋佳 (東京大学 大学院農学生命科学研究科 生産・環境生物学専攻 准教授)
発表のポイント
- 国内のリンゴ品種群において、主な起源品種のハプロタイプ(注1)の遺伝を自動的に追跡する方法を開発しました。
- ゲノムワイド関連解析(GWAS)(注2)では、果皮の着色が良いリンゴの育成に利用されてきた可能性がある起源品種のハプロタイプを明らかにしました。
- ゲノミックセレクション(GS)(注3)予測モデルの評価では、果実のリンゴ酸含量などを高い精度で予測ができ、品種改良の効率化が期待されます。
発表概要
国内のリンゴ品種は主に 7つの起源品種に由来しているため、14種類のハプロタイプが存在すると考えられます(図1)。これらのハプロタイプの遺伝を正しく追跡することができれば、国内品種が持つ性質の多様性を、より正確に理解することができる可能性があります。
東京大学、農研機構および九州大学の研究グループは、これら 14種類のハプロタイプの遺伝を自動的に追跡する手法を開発しました。この手法を用いることで、研究に供試した全リンゴ個体の 92%のゲノム領域を、 14種類のハプロタイプ情報で表すことができました。このハプロタイプ情報を利用したゲノムワイド関連解析(GWAS)では、果皮の着色が良いリンゴの育成に利用されてきた可能性がある起源品種のハプロタイプを明らかにしました。さらに、ゲノミックセレクション(GS)予測モデルの評価では、果実のリンゴ酸含量などを高い精度で予測しました。
起源品種のハプロタイプ情報は、個体の系譜情報と組み合わせて遺伝を可視化することで、リンゴの品種改良の歴史を紐解くことができます。今回新しく見いだした有望な起源品種のハプロタイプは、今後の新品種開発への利用も期待されます。
発表内容

図1 7つのリンゴ起源品種における14種類のハプロタイプ
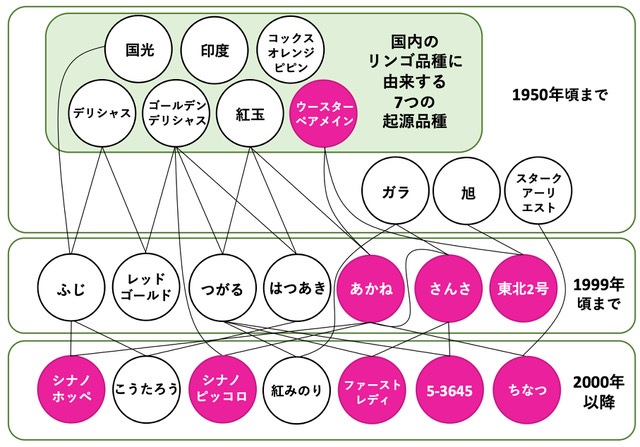
図2 リンゴの果皮の着色に関わる「ウースターペアメイン」の1つのハプロタイプの品種群における伝播
ピンクは「ウースターペアメイン」の1つのハプロタイプ(ハプロタイプ10: 図1)を有する品種を表す。
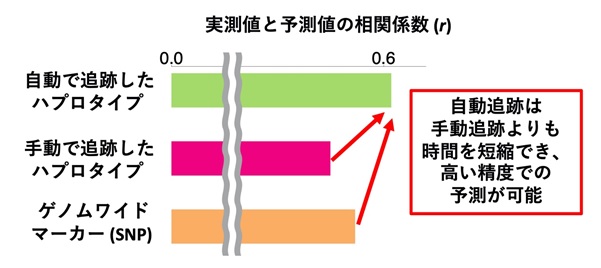
図3 果実のリンゴ酸含量のゲノミックセレクション(GS)予測モデルの予測精度
リンゴの起源品種のハプロタイプの遺伝を正しく追跡することができれば、国内品種が持つ性質の多様性の正確な理解につながると考えられますが、手動で行うことはとても困難です。そこで本研究では、コンピューターアルゴリズムを利用することで、ゲノムワイドマーカー(注4)の情報から、起源品種のハプロタイプの遺伝を自動的に追跡する方法を開発しました。また、ゲノムワイド関連解析(GWAS)と、ゲノミックセレクション(GS)予測モデルによる果実の性質の予測精度評価を行い、起源品種のハプロタイプ情報を利用する可能性についても検討しました。
農研機構で栽培・維持されている国内のリンゴ 185品種・系統と、16の交配組み合わせから育成した 659個体を供試しました。複数のコンピューターアルゴリズムを組み合わせることで、11,786個のゲノムワイドマーカーの情報から、14種類の起源品種のハプロタイプの遺伝を自動的に追跡する方法を開発しました。この方法を用いることで、使用した全リンゴ個体の 92%のゲノム領域を、14種類のハプロタイプ情報で表すことができました。手動でもハプロタイプの遺伝を追跡し、そのデータを正答として仮定した場合、自動的に追跡した方法は 90%と高い正答率を示しました。
14種類のハプロタイプ情報で表された品種群と育成個体のデータを用いて、ゲノムワイド関連解析(GWAS)を行ったところ、「ウースターペアメイン」という起源品種の1つのハプロタイプが、果皮の着色と最も強い関連を示しました。個体の系譜情報と照らし合わせて、このハプロタイプの遺伝を可視化することにより、世代が進むにつれて、このハプロタイプの頻度が集団内で有意に増加していることを明らかにしました(図2)。このハプロタイプは、果皮の着色の良いリンゴの育成に利用されてきた可能性があります。 ゲノミックセレクション(GS)予測モデルの精度評価では、果実のリンゴ酸含量を高い精度(実測値と予測値との相関係数(r)(注5)が 0.6より大きい)で予測ができました(図3)。また、自動的に追跡した起源品種のハプロタイプ情報を用いた場合、手動で追跡したハプロタイプ情報や、ゲノムワイドマーカーの情報を用いた場合よりも、高い精度で予測ができていることがわかりました(図3)。
以上の結果より、起源品種のハプロタイプ情報は、系譜情報と照らし合わせてその遺伝を可視化することにより、リンゴの品種改良の歴史を紐解くことができます。有望な起源品種のハプロタイプは、今後の新品種開発への利用も期待されます。また、起源品種のハプロタイプ情報を利用したゲノミックセレクション(GS)は、リンゴの品種改良の効率を向上できる可能性があります。
有望な起源品種のハプロタイプを組み合わせることで、生産者や消費者のニーズに対応したさまざまな新品種の開発が進むことが期待されます。今回開発した、起源品種のハプロタイプの遺伝を自動的に追跡する方法は、リンゴと同じような他殖性作物への応用も可能です。今後は、起源品種のハプロタイプ遺伝を自動的に追跡する方法の精度をさらに向上させるために、新しい材料のゲノムワイドマーカー情報を増やすとともに、ゲノムワイドマーカーの精度や適切な数についても検討を進める予定です。
なお、本研究は、JSPS科研費(JP19J40071)と、農林水産省委託プロジェクト「ゲノム情報を活用した農産物の次世代生産基盤技術の開発」の支援を受けて行われました。
発表雑誌
- 雑誌名
- Horticulture Research
- 論文タイトル
- Tracing founder haplotypes of Japanese apple varieties: Application in genomic prediction and genome-wide association study
- 著者
- Mai F. Minamikawa*, Miyuki Kunihisa, Koji Noshita, Shigeki Moriya, Kazuyuki Abe,Takeshi Hayashi, Yuichi Katayose, Toshimi Matsumoto, Chikako Nishitani, Shingo Terakami, Toshiya Yamamoto, Hiroyoshi Iwata*
- DOI番号
- 10.1038/s41438-021-00485-3
- 論文URL
- https://www.nature.com/articles/s41438-021-00485-3
問い合わせ先
日本学術振興会 特別研究員 南川 舞 (みなみかわ まい)
(東京大学 大学院農学生命科学研究科 生産・環境生物学専攻 生物測定学研究室)
Tel:03-5841-5068
Fax:03-5841-5068
E-mail:aminami<アット>g.ecc.u-tokyo.ac.jp <アット>を@に変えてください
東京大学 大学院農学生命科学研究科 生産・環境生物学専攻 生物測定学研究室
准教授 岩田 洋佳 (いわた ひろよし)
Tel:03-5841-5068
Fax:03-5841-5068
E-mail:hiroiwata<アット>g.ecc.u-tokyo.ac.jp <アット>を@に変えてください
用語解説
- 注1 起源品種のハプロタイプ
ほとんどのリンゴは二倍体ですので、2本の相同染色体が存在します(図1)。そのうちの1本は母方から、もう1本は父方から受け継いでいます。本研究では、この1本の染色体を1つのハプロタイプとして定義しています。 - 注2 ゲノムワイド関連解析(GWAS)
多数の品種・系統におけるDNAの違い(マーカー遺伝子型)と性質(表現型)の関係を数式によってモデル化し、表現型と関連するマーカー遺伝子型の違いを統計的に検出する手法です。表現型と関連するマーカー遺伝子型の違いが明らかになれば、そのマーカー遺伝子型の近傍を探索することで、表現型を制御する候補遺伝子を同定することができます。 - 注3 ゲノミックセレクション(GS)
DNAの違い(マーカー遺伝子型)の情報をもとに、個体の遺伝的能力を予測して選抜する方法です。マーカー遺伝子型と性質(表現型)のデータについて、多数の品種・系統をトレーニングデータとして両者間に見られる関係を数式によってモデル化し、その「予測モデル」に基づいて、個体の遺伝的能力を予測します。芽生えの段階で将来できる果実の性質を予測することも可能です。果樹は一般的に交配育種に長い年月を要します。また、個体サイズが大きいために多数の個体を選抜対象にできず、新品種の育成が容易ではありません。ゲノミックセレクションは、芽生えの段階で多くの優れた個体を選抜できるため、品種改良の効率を向上できる可能性があります。 - 注4 ゲノムワイドマーカー
ゲノムとは生物が生きていくために必要な遺伝情報(DNA)のセットのことです。ゲノムワイドマーカーとはゲノム全体に配置した高密度なDNAマーカーです。DNAマーカーとは生物個体の遺伝的性質、もしくは系統(個人、親子・親族関係、血統あるいは品種など)の特定に利用できる、特定のDNAの部位を目印として利用することです。個体ごとに特有のマーカー遺伝子型を持ちます。本研究では、一塩基多型(Single Nucleotide Polymorphism: SNP)をゲノムワイドマーカーとして用いています。 - 注5 相関係数
相関係数とは2つの変数間の関係の強弱を表す数値です。本研究では、0〜1の範囲の値で、実測値と予測値の相関の強弱を表しています。数値が1に近いほど強い相関があると考えられ、ゲノミックセレクション予測モデルによる予測値が実測値と一致していることを表しています。

