膨大な文献データをつなぎ合わせて新たな関連性を導出する手法を開発。 COVID-19関連薬探索へ応用。
- 発表者
- 村松 知成 (東京大学大学院農学生命科学研究科 附属食の安全研究センター 特任准教授)
田之倉 優 (東京大学大学院農学生命科学研究科 特任教授)
発表のポイント
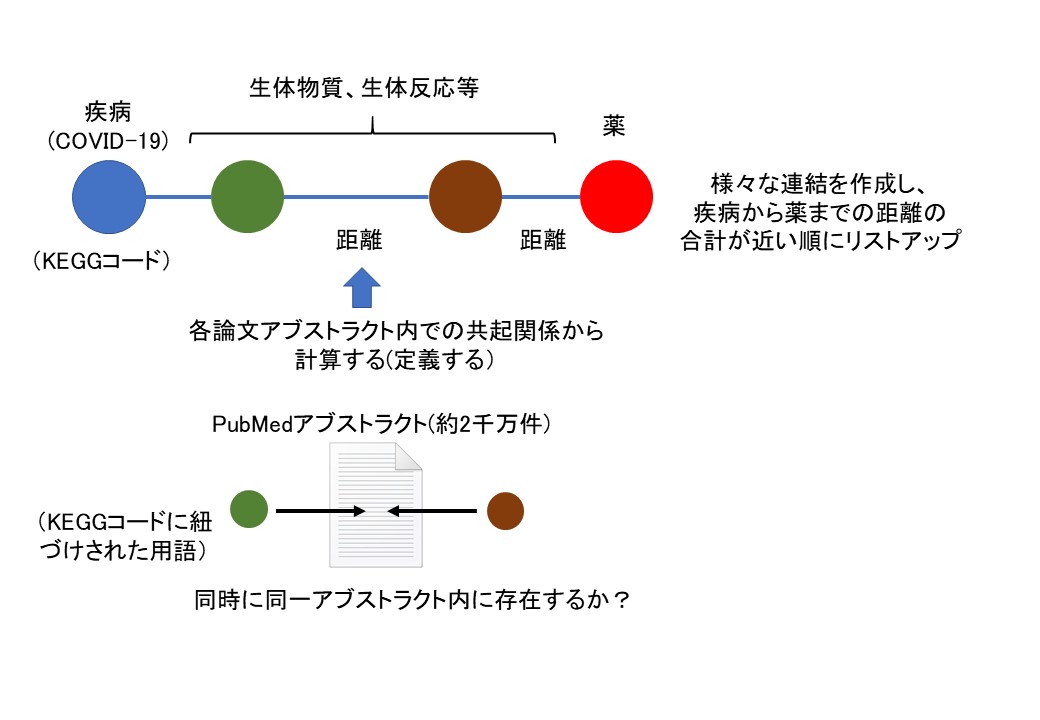
- 利用可能な文献アブストラクト(約2千万件)を解析し、様々な生体分子や生命現象間の関連性を導出、連結することにより新たな関連性を推定するシステムを開発しました。
- このコンピュータープログラムは、研究者が関連論文を読んでそれぞれの関連性をつなぎ合わせながら新規の関連性を推定する方法を模した手順で行うところに新規性があります。
- 今回はCOVID-19関連薬の推定に応用し検証を行いましたが、どのような生体分子、生命現象についての関連性の推定にも適用可能です(他の疾病関連薬の推定など)。
発表概要
深刻な感染症COVID-19の収束には特効薬が望まれますが、新薬の開発・実用化には時間が必要です。代替法として他の疾病に対する既存薬の転用も検討されていますが、莫大な化合物が対象となるため、先立つ論理的検討により候補を絞り込むことが効率的です。この目的のため、東京大学大学院農学生命科学研究科の村松知成特任准教授と田之倉優特任教授はCOVID-19の治療薬候補を特定するための新しく強力なツールを開発しました。これは生体内における各種反応、現象、物質などの相互関連性に基づきます。文献データベースPubMedと生体内における各種反応、現象、物質を扱うKEGGという2つの巨大なデータベースを用い、PubMedの要約フィールドを解析することにより、21,589,326件のPubMed IDと98,556件のKEGGコードを関連付け、それから各KEGGコード間の関連性を導き出し、それらを繋いでいくことにより新たな関連性を推測します。今回はこの方法によりCOVID-19関連薬の推定を行いましたが、このプログラムは他の感染症にも適用でき、将来新しい感染症が出現した場合にも迅速な薬剤候補特定に役立ちます。
発表内容
地球規模で猛威をふるう新型コロナウイルスに対し現在最も有効な手立てとしてはワクチン接種がありますが、将来的には同じワクチンが有効であるとは考えられません。また、ワクチンは基本的に予防的手段であり、感染者に対する根本的な治療薬の必要性は依然として存在しています。ところがCOVID-19に対しては、インフルエンザに対するタミフルのような強力な特効薬は現状では存在しません。新薬の開発と実用化には長い時間が必要なのでその代替法として他の疾病に対する既存薬の転用(ドラッグリポジショニング)が検討されてきています。これを効果的に行うためには実験によるスクリーニングに先立ち、インシリコスクリーニング等コンピューターを用いた絞り込みが有効であると考えられますが、これにも数量的な限度はあります。また、これらの前段階スクリーニングは、作用機序を想定して行い、仮に実際には効いたとしても想定された作用機序と異なるものは見過ごされることになります。
これに対し、今回、研究グループはPubMedという膨大な文献データベースをすべて用いてCOVID-19関連薬を探索・推定しました。これは、PubMedデータベースに含まれアブストラクト項目の存在する約2千万件のレコードについて、そのアブストラクト部分での用語の共起に基づきます。COVID-19, SARS-CoV-2等当該疾病に関係する用語と薬(既存薬)に関する用語の共起を調べ、それが存在するPubMedレコード(PubMed ID)の数から算出した確からしさを評価するだけでなく、未だ報告のない関連薬の推定をも意図しています。
KEGG(Kyoto Encyclopedia of Genes and Genomes)という生体内における各種反応、現象、物質を扱うデータベースから抽出したすべての用語(疾病、薬以外も含む)間の関連性を、各PubMedアブストラクト項目での共起に基づき解析しました。各用語は抽出元となった各KEGGコードに紐づいているためKEGGコード間の関連性を得ることができます。
新たな関連性の推定については、次のように行います。新たな関連性の推定は三段論法的な考え方をします。すなわち、関連性の大きなものどうしをつないでいくのです。まず、KEGGコード間の距離(関数)を定義し、COVID-19(H02398)を起点に近いものを順次つないでいくことにより「木」構造を作成、その中に現れた薬コード(Dコード)をそれに至る距離の合計が小さい順に拾っていきます。
この方法の検証は次のように行いました。COVID-19とPubMedアブストラクト内で直接共起の見られた薬剤について、その直接共起情報のみをすべて除いてコードをつないで行くことにより得られる間接的な距離と、直接見られた共起から計算される距離を比較します。近い距離のものからリストアップすると上位よりレムデシビル、ヒドロキシクロロキン、アビガン等を先頭に妥当と思われる順で関連薬が並び、また、それらの中では間接距離と直接距離の良い一致がみられることから、方法の有効性が確認されました。逆に、PubMedアブストラクト内での直接共起が見られないにもかかわらず、比較的近い距離で間接的につながる薬もあり、これらは新薬候補とすることができます。
今回開発したプログラムの特徴としては以下のものを上げることができます。1) COVID-19関連薬探索のみならず、他の任意の疾病に対する薬の探索・推定にも用いることができる。特に、将来、新たな感染症が出現した場合、初期段階では、その感染症では直接的な薬の報告がない状態となるが、その場合にも関連薬候補をリストアップすることができる。2) さらに、疾病-薬関係だけではなく、あらゆる生命現象間の関連性の抽出・推定にも用いることができる。3) 用いている距離の計算は用語どうしのPubMedアブストラクト内での共起に基づく。したがって、今後、この部分の改良を行うことによってさらに精度の高い予測を行えるようになる可能性がある。4) すべてのデータはPubMed IDに紐づけされており、容易に元論文を確認することができる。5) PubMedアブストラクトからの用語の抽出には一般的に自然言語処理で用いられるステマー(すべてを小文字化し、語幹のみを抽出するプログラム)を医学や生物学で多用される語尾(-ase, -ose, -ine, -sisなど)や大文字略語(DNA、SARSなど)に対応できるように改変して用い、文献マイニングとしての情報抽出精度の向上も図っている。
本研究は、国立研究開発法人日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム(BINDS)(課題番号P20am0101068)の支援を受けて行われました。
発表雑誌
- 雑誌名
- Bioinformatics Advances
- 論文タイトル
- A novel method of literature mining to identify candidate COVID-19 drugs
- 著者
- Tomonari Muramatsu*, Masaru Tanokura*
- DOI番号
- 10.1093/bioadv/vbab013
- 論文URL
- https://academic.oup.com/bioinformaticsadvances/advance-article/doi/10.1093/bioadv/vbab013/6325500
問い合わせ先
東京大学大学院農学生命科学研究科 附属食の安全研究センター
特任准教授 村松 知成(むらまつ ともなり)
Tel:03-5841-2279
E-mail:atmuramatsu<アット>g.ecc.u-tokyo.ac.jp <アット>を@に変えてください。
東京大学大学院農学生命科学研究科
特任教授 田之倉 優(たのくら まさる)
Tel:03-5841-5165
E-mail:amtanok<アット>g.ecc.u-tokyo.ac.jp <アット>を@に変えてください。