機械学習を用いたタンパク質ペアの 酵素触媒反応の同一性予測手法の開発
発表のポイント
- 2つのタンパク質間の配列と構造の類似度を用いて、2つのタンパク質が同じ酵素反応を触媒するかどうかを予測する手法FUJISANを開発しました。
- FUJISANは配列の類似度のみを用いた手法や最先端のディープラーニングモデルを用いた手法よりも高い予測性能を示しました。
- FUJISANを用いると、様々な生物の膨大なゲノム情報の中から、有用な反応を触媒する新規タンパク質を効率的に探索することが可能になると期待されます。
概要
東京大学大学院農学生命科学研究科の藤田卓特任研究員と、寺田透教授らによる研究グループは、タンパク質の配列と構造の類似度を解析することにより、2つのタンパク質が同じ酵素反応を触媒するかどうかという機能を予測する手法(FUJISAN)を開発しました(図1)。
FUJISANの予測性能は全長配列類似度のみに基づくモデルや、最先端のディープラーニングモデルなどの既存のアプローチを凌駕しました。タンパク質の機能を実験的に決定することは、コストが高く、時間もかかるため、FUJISANを用いたタンパク質の機能を予測する計算手法は、今後のタンパク質機能に関わる研究全般に役立つことが期待されます。
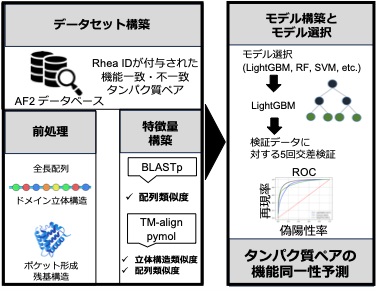
図1:本研究のワークフロー
発表内容
これまでの先行研究では、タンパク質の機能を担う配列は進化的に保存されているという経験的な知見をもとに、研究者はBLASTp(注1)のような配列類似度検索プログラムを用いて、機能既知のタンパク質と比較することによって、対象のタンパク質の機能を予測してきました。しかし、アミノ酸配列のわずかな変化が基質特異性に影響を与える可能性があるため、これらのプログラムは酵素の機能、特に基質や生成物の予測にはあまり有効ではありません。
この度、本研究チームは深層学習ベースのタンパク質構造予測プログラムAlphaFold2(AF2)のデータベースに登録されている予測構造を用いて、配列情報に加えて立体構造情報を用いて酵素反応レベルの機能予測を試みました。機能のラベルとして、Rhea ID(注2)を用いました。AF2の予測構造の中でも相対位置が正確なドメイン構造(注3)や、酵素反応を触媒する際に重要であるポケット形成残基(注4)を抽出し、タンパク質のペアごとに特徴量を計算しました(全長配列間の類似度、ドメイン間の構造・配列類似度、ポケット間の構造・配列類似度)。
既存の機械学習モデルの中からモデル選択(注5)と検証データに対する5回交差検証(注6)を通して、最も予測性能が高いモデルを決定し、FUJISANと名付けました。FUJISANの予測性能を既存の手法と比較すると、いずれの手法よりもAUROC(Area Under the ROC curve)値(注7)が高いことがわかりました(図2左)。AUROCが高いほど予測性能が高いことを示すため、FUJISANは比較した既存手法の中で最も予測性能が高いことを意味します。
FUJISANの各特徴の分類に対する有効性を評価するため、ジニ重要度(注8)を計算しました。解析の結果、「ドメイン間の配列類似度」が最も影響力のある特徴であることが明らかになりました(図2右)。したがって、配列特徴よりも立体構造特徴の方が予測には重要であることを直接的に示しました。様々な生物種のゲノム配列が決定されている一方で、タンパク質の多くはその機能が未解明のままとなっています。本研究成果は、このようなタンパク質の機能解析を加速するだけでなく、有用な反応を触媒する新規タンパク質の発見に寄与すると期待されます。
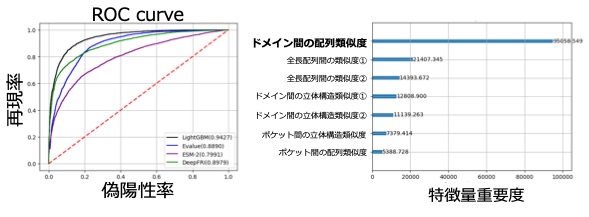
図2:FUJISANの予測性能(左)と特徴量のジニ重要度(右)
左:各手法のROCcurveの比較(括弧内は各手法のAUROC値)。右:ジニ重要度の特徴量間の比較。
発表者・研究者等情報
東京大学
大学院農学生命科学研究科 応用生命工学専攻
寺田 透 教授
藤田 卓 特任研究員(現:応用動物科学専攻 助教)
論文情報
- 雑誌
- Computational and Structural Biotechnology Journal
- 題名
- Enhanced prediction of protein functional identity through the integration of sequence and structural features.
- 著者
- Suguru Fujita, Tohru Terada*
- DOI
- 10.1016/j.csbj.2024.11.028
- URL
- https://www.csbj.org/article/S2001-0370(24)00399-4/fulltext
研究助成
本研究は、日本学術振興会の科学研究費助成事業・学術変革領域研究(A)「生体反応の集積・予知・創出を基盤としたシステム生物合成科学(予知生合成科学)」(JP22H05126)および日本医療研究開発機構(AMED)の生命科学・創薬研究支援基盤事業(BINDS)(JP24ama121027)の支援により実施されました。
用語解説
- 注1 BLASTp
アミノ酸配列からなる入力配列と相同性を持つアミノ酸配列をデータベースから検索するプログラムのこと。 - 注2 Rhea ID
酵素反応を基質と生成物によって分類しているデータベースが割り当てている、反応ごとに固有のIDのこと。 - 注3 ドメイン構造
タンパク質の中で、立体構造上独立した領域、およびその領域が作る構造のこと。 - 注4 ポケット
タンパク質の分子表面の形状において,主に低分子基質結合部位候補となるような凹領域のこと。 - 注5 モデル選択
データを予測する複数のモデルの中から、ある指標(例えば、予測性能など)に対して最も良いモデルを選ぶ手法のこと。 - 注6 5回交差検証
モデルの性能評価評価においてより信頼性の高い結果を得るための手法のこと。 - 注7 AUROC値
予測性能指標の一つ。ROC曲線下面積のこと。この指標は、0.0(=0%)~1.0(=100%)の範囲の値を取り、1.0に近いほどモデルの予測性能が高いことを示す。 - 注8 ジニ重要度
モデルが予測をする際にそれぞれ特徴量がどれだけ予測に寄与しているのかを評価する指標のこと。
問い合わせ先
東京大学大学院農学生命科学研究科 応用生命工学専攻
教授 寺田 透(てらだ とおる)
E-mail:tterada[at]bi.a.u-tokyo.ac.jp
※上記の[at]は@に置き換えてください。
関連教員
寺田 透
藤田 卓

