ゲノムの「暴れ者」トランスポゾンを押さえ込むしなやかな戦略 ――piRNAの増幅経路が持つもう一つの役割――
発表のポイント
- トランスポゾンを抑制するpiRNAの配列パターンは完全に決まったものではなく、確率的に変動しうることを発見しました。
- piRNAの配列パターンは、再生産するpiRNA同士の競争によって自動的に最適化されることを発見しました。
- トランスポゾン制御メカニズムの理解を深め、遺伝子工学や生物医学分野への応用につながる可能性があります。

雑誌の表紙:この作品は「打蛇打七寸(物事を行うにはその急所をつかなければならない)」という中国のことわざから着想を得ており、piRNAの配列パターンが自動的に最適化されることの重要性を表した絵です。
研究概要
東京大学定量生命科学研究所の泊幸秀教授と東京農工大学大学院農学研究院生物システム科学部門の庄司佳祐准教授を中心とする研究チームは、生殖細胞でトランスポゾンを抑制する役割を担うpiRNAの配列パターン(注1)が、抑制すべきトランスポゾン配列に応じて自動的に調整されていくメカニズムを明らかにしました。この自動的な調整は、カイコをはじめとして、マウスやハエなど、piRNA増幅機構であるピンポン経路を持つ生き物で広く観察されました。この仕組みは、変化し続けるトランスポゾンをpiRNAが永続的に抑制し続けるための柔軟な仕組みであると言えます。
研究内容
【背景】
生物を形づくり、生命現象を維持するためのタンパク質の機能に関する情報は、各細胞のゲノムDNAに書き込まれています。この生物の設計図とも言えるゲノムDNAは、実は半分程度がトランスポゾンと呼ばれる、勝手に増殖し、ゲノム中を移動するDNA配列によって占められています。無秩序なトランスポゾンの増殖はゲノムDNAに書き込まれている遺伝子構造の破壊をもたらします。特に、次世代に伝わる生殖細胞での遺伝子構造の破壊は、子孫への永続的な影響をもたらします。私たちが研究しているpiRNA(PIWI-interacting RNA)は、23~30塩基程度の小さなRNAですが、動物の生殖細胞内でトランスポゾンの転移を抑制し、ゲノムの安定性を維持する重要な役割を担っています。
それぞれのpiRNAは、特定のトランスポゾンの一部に対応した配列を持っており、PIWIと呼ばれるタンパク質と複合体を作ることで、そのトランスポゾンを認識して抑制します。細胞内には数多くの種類(配列パターン)のpiRNAが存在するため、ゲノム中に存在する様々なトランスポゾンを抑制することができるのです。一方で、トランスポゾンがpiRNAによる抑制を回避するには、piRNAによって認識される部分の配列を変異させることでpiRNAによって認識されにくくするという方策がありえます。ゲノムDNAに付属して遺伝してしまうトランスポゾンとの付き合いは、世代を超えた永続的なものなので、このトランスポゾンの変異戦略に対応してpiRNAが永続的にトランスポゾンを抑制できる仕組みが必要なはずです。しかし、その仕組みは不明でした。
もう一つ不明だったのは、piRNAの増幅を担う「ピンポン経路(注2, 図1)」と呼ばれるプロセスが進化的に保存されている意義です。ピンポン経路では、piRNAとPIWIタンパク質がトランスポゾンRNAを認識して切断することでトランスポゾンを抑制します。この時、切断された断片はそのまま分解されるのではなく、別のPIWIタンパク質に取り込まれて新しいpiRNAとなります。このようにして、同じ配列のpiRNAを再生産し、piRNAを増幅するシステムが、piRNAを持つ動物の間では広く保存されています。しかし、ピンポン経路は数十の遺伝子や、細胞内の複数の領域が関与する、piRNAの量を増やすためだけにしてはあまりにも複雑なシステムであり、それが多くの生物に共通して保存されている意義は不明でした。
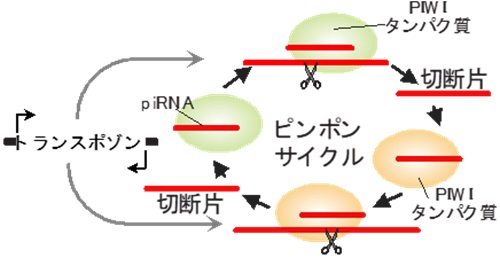
図1:piRNAの増幅経路:ピンポン経路
piRNAがPIWIタンパク質と結合し、相補的なトランスポゾンRNAを切断したのち、切断片が別のPIWIタンパク質に取り込まれることで、新たなpiRNAが生成される。その新たなpiRNAも同様にpiRNAの産生を誘導することで、最初と同じ配列のpiRNAが再生産される。
【研究成果】
研究チームは、piRNAを産生しているカイコ培養細胞を用いて、7年間の継代培養の前後で、piRNAの配列パターンを詳細に比較しました。その結果、piRNAの配列パターンは時間の経過とともに大きく変化することが明らかになりました。このことは、piRNAの配列パターンは決まったものではなく、複数のパターンの間で確率的にゆらいでいる可能性を示唆します。次に、この結果が、培養細胞でだけ起こる人工的なものでないことを確かめるため、新たに開発した手法を用いてカイコの1卵ごとのpiRNAの配列パターンの違いを解析しました。カイコの卵のpiRNAは母親によって作られたものが受け渡されるため、基本的には同じ母親由来の卵においては同一の配列パターンが観察されるはずです(図2)。しかし、実際に解析してみると、piRNAの配列パターンには個々の卵ごとに個体差が見られることが分かりました。つまり、母親が作ったpiRNAの分配は確率的な現象であり、それによって個体差が生じうることを示唆します。これらのことは、piRNAの配列パターンというものは、確率的にゆらいでおり、柔軟に変更可能であることを意味します。
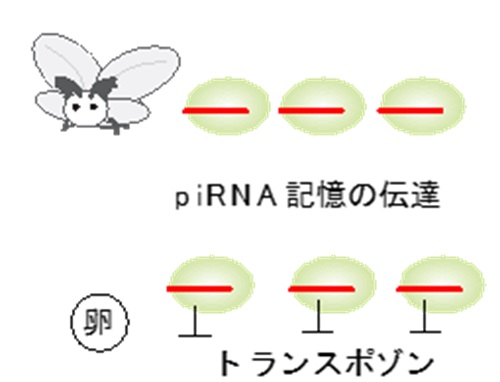
図2:世代を超えたpiRNAの伝達
カイコやキイロショウジョウバエにおいては、piRNAは卵を通じて次世代に伝達される。伝達されたpiRNAはトランスポゾンを抑制し、その断片からpiRNAを再生産する。piRNAは抑制すべきトランスポゾンの記憶を次世代に伝える役割も持つ。
次に、数理モデルを用いた解析により、隣接するpiRNA同士の競争がpiRNAの配列パターンを最適化していることを突き止めました(図3)。集団遺伝学の文脈では、遺伝子を再生産する複数の生物が餌や住処で競合する際には、子孫を残す効率が高いものが自然と選抜されます。同様に、ピンポン経路によって再生産するpiRNAが材料となるトランスポゾンRNAで競合する時、piRNAの産生効率が高いものが自然と選抜されるのです。この仕組みの利点は、piRNAが適しているかを一々判断することなく、ピンポン経路によるpiRNAの再生産と競合を続けることで、より適したpiRNAの配列パターンを自然に得ることができる点です。そのため、標的トランスポゾンが変異してpiRNAの抑制効率が低下しても、自動的にpiRNA配列パターンを調整して抑制効率を上げることで、長期的なトランスポゾン制御を達成していると考えられます。また、このような現象は、ピンポン経路を持つキイロショウジョウバエやマウスでも確認され、piRNAに普遍的なメカニズムであることが分かりました。つまり、複雑なピンポン経路は、piRNAの増幅とともに、piRNA配列パターンの自動的な最適化のためであったと言えます。
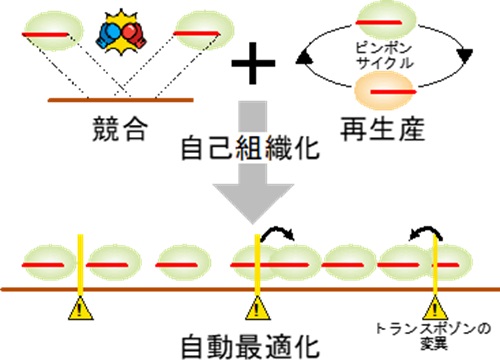
図3:piRNA配列パターンの自動的な最適化
同じ材料(同じトランスポゾン)を由来とするpiRNA同士は競合する。これに、ピンポン経路によるpiRNAの再生産を組み合わせると、piRNAの配列パターンが自動的に最適化される。例えばpiRNAによる認識領域にトランスポゾンの変異が起こり、再生産の効率が低くなる一方で、競合する別のpiRNAには影響がない場合を仮定する。その場合、再生産の複数サイクルの後に、効率が低くなったpiRNAは失われ、効率が低下していない場所からつくられる競合piRNAに置き換わる。
【今後の展開】
本研究の成果は、「トランスポゾンの変異戦略に対応してpiRNAが永続的にトランスポゾンを抑制できる仕組み」についての重要な手がかりを提供します。本研究に基づくと、最適化されたpiRNAの配列パターンが卵を通じて次世代に伝わり、さらに次代で最適化されることで、世代を追うごとにより最適な配列パターンが実現されていくと考えられます。卵におけるpiRNAの配列パターンのゆらぎは、個体間の多様性を生み出し、その中でトランスポゾン抑制に適した特徴を持つ個体が生存しやすくなることで、集団全体の適応度を高める役割を果たすと考えられます。実際には、今あるトランスポゾンに対しては、すでに最適な配列パターンが実現していると考えられるので、今後は、実際に高頻度に変化するトランスポゾンや、新規のトランスポゾンに対してpiRNAの配列パターンがどのように変化していくのかを捉えていく必要があります。
また、本研究の結果、piRNAの配列パターンは、確率的に変動しうることが判明しました。そのため、例えば疾患と関連するpiRNAが見つかった際も、その関連がただのゆらぎによるものではないのかを吟味する必要が出てきたと言えます。さらに、本研究で明らかになったpiRNAの自律的最適化メカニズムは、トランスポゾン制御を応用した遺伝子工学や生物医学分野への発展にも寄与する可能性があります。
〇関連情報:
プレスリリース
「piRNAはどのようにつくられるのか?
~「Zucchini(ズッキーニ)」によるRNA切断ルールの解明に成功 ~」(2020/01/30)
https://www.iqb.u-tokyo.ac.jp/pressrelease/200130/
発表者
東京大学
定量生命科学研究所 附属高度細胞多様性研究センター RNA機能研究分野
泊 幸秀 教授
喩 婕(ユ ジエ) 技術補佐員
河﨑 史子 助教
泉 奈津子 技術専門職員
大学院農学生命科学研究科 生産・環境生物学専攻
勝間 進 教授
木内 隆史 准教授
東京農工大学
大学院農学研究院 生物システム科学部門
庄司 佳祐 准教授
論文情報
- 雑誌
- Molecular Cell
- 題名
- Autonomous Shaping of the piRNA Sequence Repertoire by Competition between Adjacent Ping-Pong Amplification Sites.
- 著者
- Jie Yu, Fumiko Kawasaki, Natsuko Izumi, Takashi Kiuchi, Susumu Katsuma, Yukihide Tomari*, Keisuke Shoji* (* 共同責任著者)
- DOI
- 10.1016/j.molcel.2025.02.015
- URL
- https://doi.org/10.1016/j.molcel.2025.02.015
研究助成
本研究は、科研費「基盤研究(S): 反応場に着目したpiRNA経路の生化学的解析(課題番号:18H05271)」、「基盤研究(A): piRNA配列レパートリーの決定と継承:トランスポゾンを永続的に抑制する巧妙な戦略(課題番号:23H00364)」、「若手研究: piRNAセンサーシステムを用いたpiRNAクラスタの成立条件の解明(課題番号:22K15082)」、「学術変革領域(A): 細胞質共生細菌ボルバキアのゲノム編集による性制御と共生機構の解明(課題番号:24H02278)」、「基盤研究(B): 細胞内共生細菌による宿主piRNA 経路操作機構の解明(課題番号:24K01769)」の支援により実施されました。
用語解説
- 注1)piRNAの配列パターン:
piRNAの配列の多様性や組み合わせを指す。 - 注2)ピンポン経路:
piRNAの増幅機構の一つで、二つのPIWIタンパク質が関与する。piRNAがPIWIタンパク質と結合し、相補的なトランスポゾンRNAを切断したのち、切断片が別のPIWIタンパク質に取り込まれることで、新たなpiRNAが生成される。この過程が繰り返されることにより、トランスポゾン由来のRNAが効果的に抑制されるとともに、特定のpiRNAの配列が増幅される。(図1も参照ください)。
問い合わせ先
(研究内容については発表者にお問合せください)
東京大学 定量生命科学研究所 高度細胞多様性研究センター RNA機能研究分野
教授 泊 幸秀(とまり ゆきひで)
FAX: 03-5841-8485
E-mail: tomari[at]iqb.u-tokyo.ac.jp
東京農工大学 大学院農学研究院 生物システム科学部門
准教授 庄司 佳祐(しょうじ けいすけ)
TEL: 042-388-7921
E-mail: kshoji[at]go.tuat.ac.jp
東京大学定量生命科学研究所総務チーム
TEL: 03-5841-7813
E-mail: soumu[at]iqb.u-tokyo.ac.jp
東京農工大学 総務課広報室
TEL: 042-367-5930
E-mail: koho2[at]cc.tuat.ac.jp
※上記の[at]は@に置き換えてください。

