微生物群と代謝物のデータを統合し分類する新解析法I-SVVSを開発
発表のポイント
- 微生物叢とホスト生物の相互作用を理解し、健康や環境への影響を解明するための新しい解析手法として、高次元マルチオミクス・マイクロバイオーム解析ツールを開発しました。
- 微生物叢の配列データと代謝物データを統合解析する統合確率的変分変数選択(I-SVVS)という新規手法を提案し、従来よりも高精度なクラスタリングを実現しました。
- これは、サンプルごとに異なる特徴を抽出し、代謝物と微生物の種類の関係に新たな知見を提供するもので、確率的変分推論を活用することで、計算負担を大幅に軽減し、大規模データ解析にも対応できるようになりました。
マルチオミクス・マイクロバイオームのためのI-SVVS法
概要
東京大学大学院農学生命科学研究科の岩田洋佳教授らは、統合確率的変分変数選択(I-SVVS)という新しい手法を開発しました。これは、植物の根圏土壌や動物の腸内など、ホスト生物の近傍に存在する微生物叢(マイクロバイオーム)と代謝物(メタボローム)のデータを統合し、分類(クラスタリング(注1))を行うことで、微生物と代謝物の関係を解明する技術です。微生物と代謝物の相互作用は、ホスト生物の成長や健康、さらには環境に影響を与えるため、その仕組みを理解することが重要です。
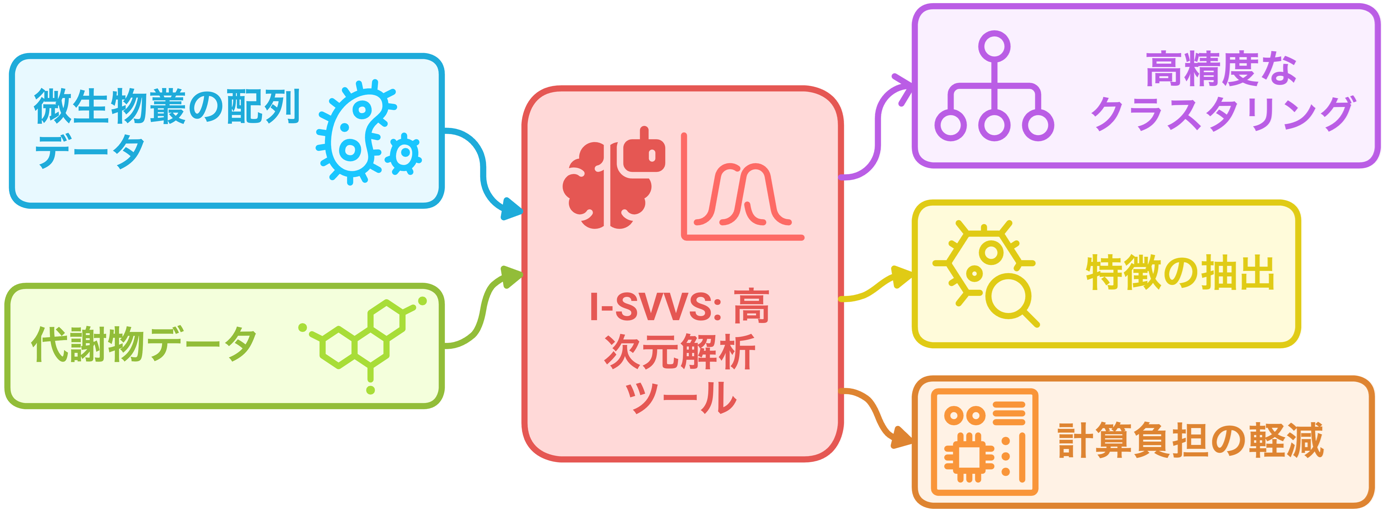
I-SVVSは、ベイズ非パラメトリック手法(注2)を用いることで、最適な分類群(クラスタ)数を自動的に決定し、データの特徴に基づいた正確な分類を可能にします。また、変数選択の仕組みにより、分析対象のデータから特に重要な特徴を特定することができます。さらに、確率的変分推論(注3)によって計算負担を軽減し、大規模なデータセットの解析を高速かつ効率的に行うことができます(図1)。
この手法により、多様なホスト生物における微生物叢と代謝物の相互作用をより深く理解することが可能となり、農業・医療・環境科学の発展に貢献することが期待されています。
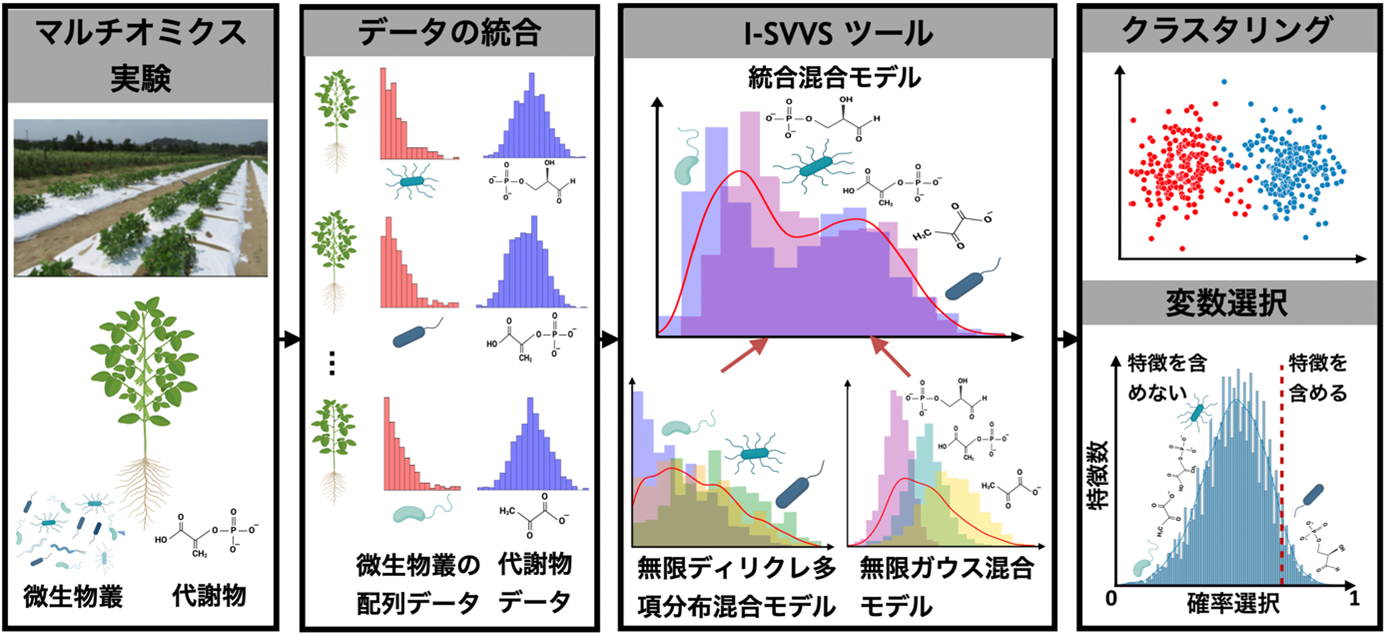
図 1:I-SVVS手法の概要
発表内容
マルチオミクス・マイクロバイオーム(微生物叢)データとは、微生物群の働きや相互作用を理解するために、メタゲノム(微生物種を表す遺伝情報)やメタボローム(代謝物)など、さまざまなオミクス技術を統合したデータです。しかし、このデータは情報量が多い一方で、個々のデータ点の密度が低く、また複数の変数が強く関連し合う(多重共線性)という特徴があり、正しく解析するためには高度な計算手法が必要とされます。
この課題を解決するため、私たちは統合確率的変分変数選択(I-SVVS)と名付けた統計手法を開発しました。これは、高次元のマイクロバイオームデータの解析に特化した初の手法です。I-SVVSは、確率的変分推論に基づく変数選択の枠組みを採用し、微生物データには「無限ディリクレ多項分布混合モデル(注4)」、代謝物データには「無限ガウス混合モデル(注5)」を適用することで、計算効率と解析精度を向上させました。さらに、データ内の膨大な特徴の中から、最も重要な情報を持つ最小限の特徴セットを特定し、高精度な分類(クラスタリング)と微生物-代謝物の関連性の発見を可能にしました。
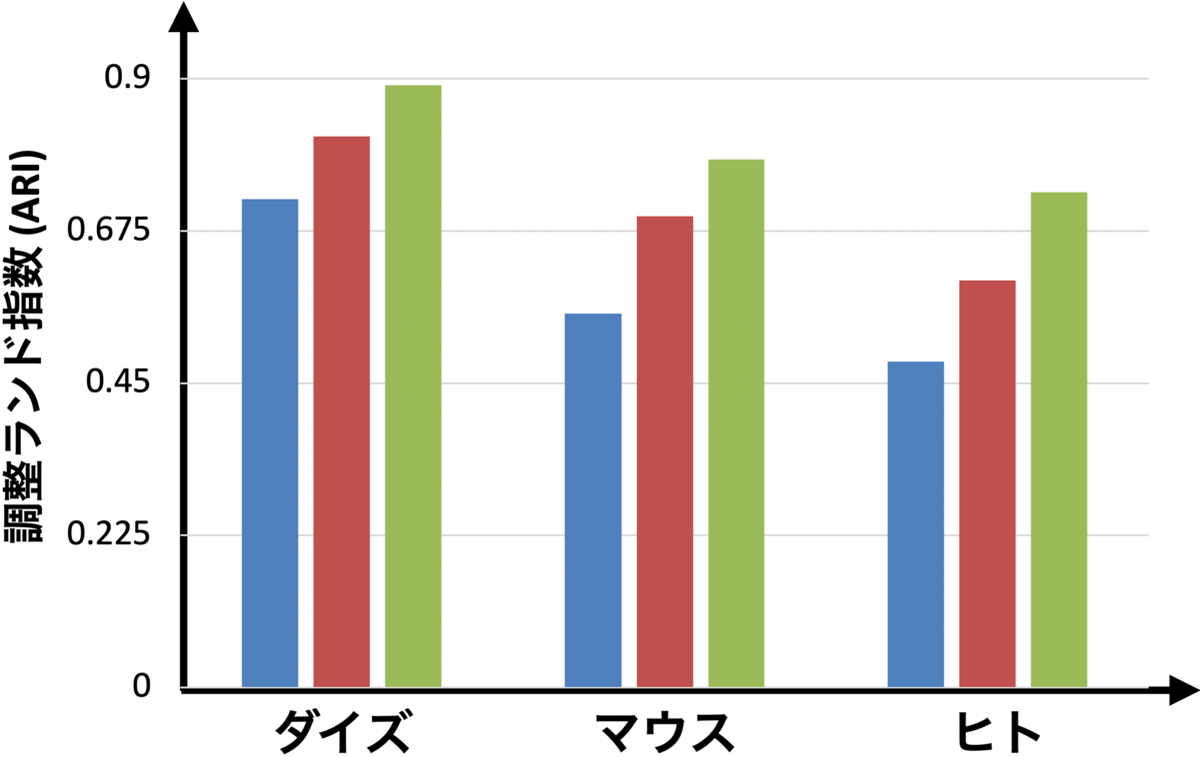
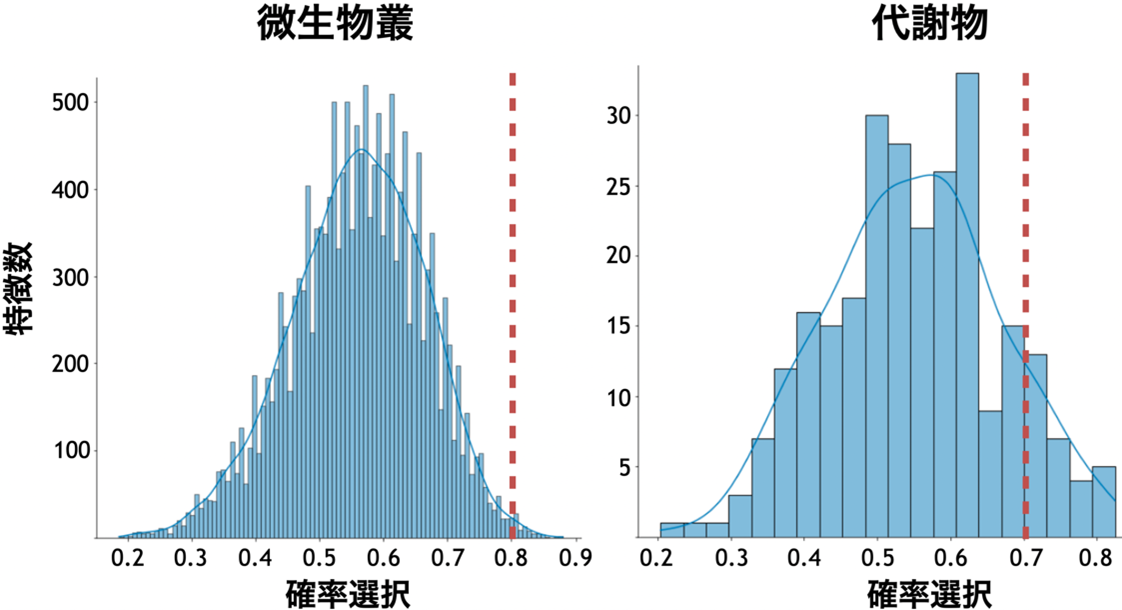
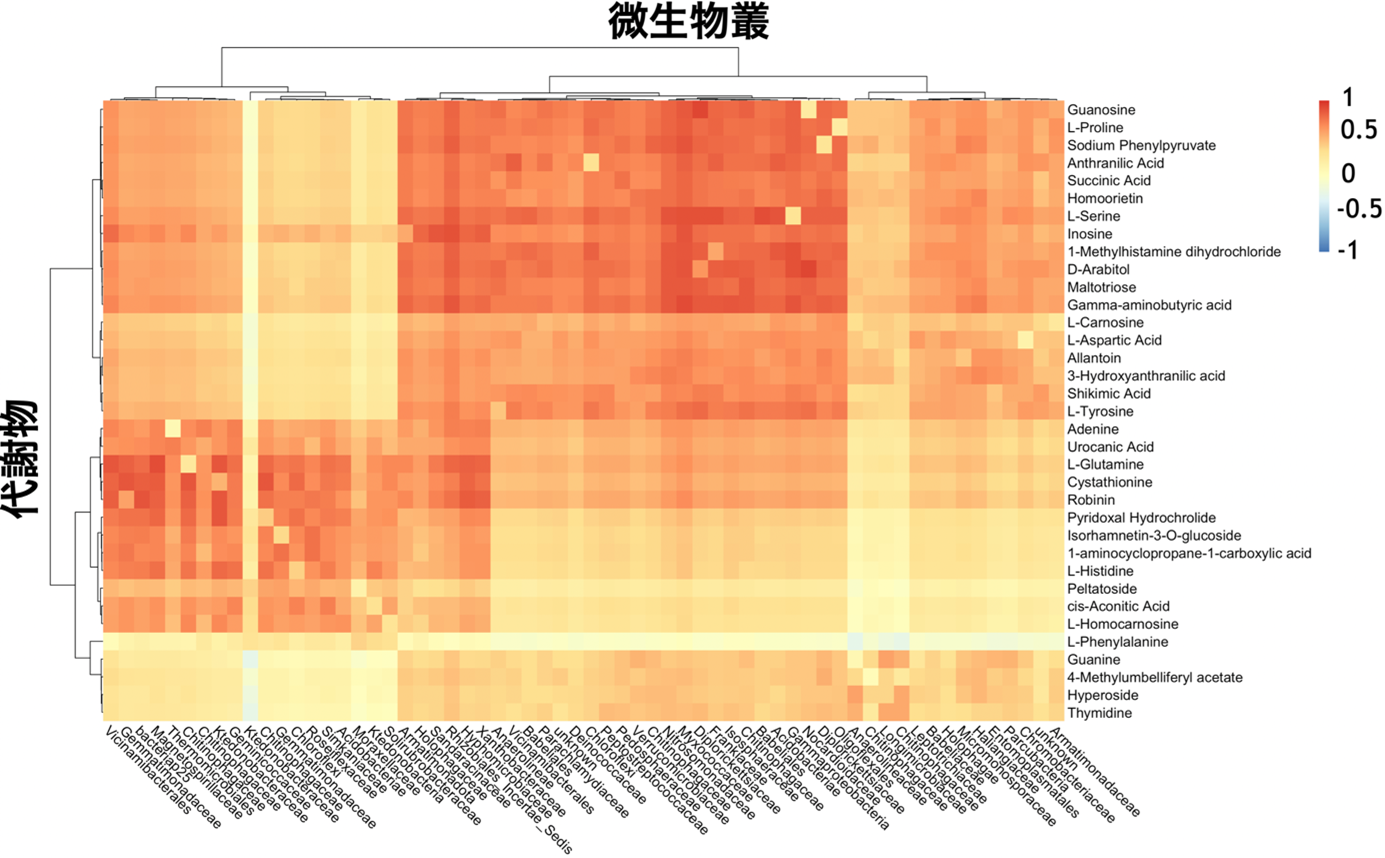
I-SVVSの性能を検証するため、ダイズ・マウス・ヒトの3つのデータセットを用いて実験を行いました。その結果、すべてのケースで、従来の解析手法(iClusterPlus(注6)やClusternomics(注7))よりも高い精度と高速な処理能力を示しました。特に、ダイズのデータセット(377サンプル、16,943種の微生物、265の代謝物)では、I-SVVSはより正確で素早い解析が可能であることが確認されました(図2)。さらに、ダイズの生育と関連する重要な微生物群を特定し(図3,4)、さまざまな環境条件下での微生物-代謝物の関連性も明らかにしました(図5)。
これらの結果は、I-SVVSがマルチオミクス・マイクロバイオーム研究を大きく前進させる可能性を持つことを示しています。これまで微生物の働きとホスト生物の健康・成長の関係を明確に捉えることは難しく、複雑なデータを解析するためには時間や労力がかかっていました。しかし、I-SVVSを活用することで、微生物がどのようにホスト生物と相互作用し、成長を助けたり病気を引き起こしたりするのかをより正確に理解できるようになります。たとえば、土壌中の微生物がどのように植物の成長を促進するのか、腸内細菌が人間の健康にどのような影響を与えるのかを詳細に解析することが可能になります。これは、農業分野では作物の収量向上や持続可能な農業の推進に、医療分野では腸内細菌を活用した疾患予防や治療法の開発につながる可能性があります。環境分野においても、微生物が生態系のバランスに果たす役割をより深く理解し、自然環境の保全や気候変動への対応に貢献できると考えられます。
今後、I-SVVSを活用することで、微生物とホストの関係をより深く解明し、農業・医療・環境科学といった幅広い分野での応用が期待されます。
図 2:ダイズデータに対する3つのアプローチのARIスコア。
青色バーが Clusternomics、赤色バーが iClusterPlus、緑色バーが I-SVVS。
図 3:ダイズデータにおける微生物叢と代謝物の選択に関する事後分布の平均。
破線は微生物叢と代謝物プロファイルの特徴を選択するための境界線。

図 4: 統合確率的変分変数選択(I-SVVS)アプローチを用いて選択され、ダイズデータの系統樹上にマッピングされた微生物種。
赤色のプラス記号はコントロールを、青色の星印は干ばつを示す。
図 5:微生物叢と代謝物のヒートマップ法は、ダイズデータについて、I-SVVSアプローチによって2つのグループに選択された偽クラスタを生成した。
発表者・研究者等情報
東京大学大学院農学生命科学研究科
Dang Tung 研究当時:博士課程
現:東京大学大学院理学系研究科生物科学専攻生物情報科学科 特任研究員
戸田 悠介 研究当時:特任研究員
現:農研機構 農業環境研究部門 任期付研究員
櫻井 建吾 研究当時:博士課程
岩田 洋佳 教授
鳥取大学乾燥地研究センター
山崎 裕司 研究当時:特命助教
現:東京大学大学院農学生命科学研究科 助教
辻本 壽 研究当時:教授
現:鳥取大学 国際乾燥地研究教育機構 特任教授
理化学研究所
バイオリソース研究センター 植物-微生物共生研究開発チーム(研究当時)
熊石 妃恵 研究当時:テクニカルスタッフ
臼井 絵里香 研究当時:テクニカルスタッフ
小堀 峻吾 研究当時:開発研究員
佐藤 匠 研究当時:特別研究員
成川 恵 研究当時:開発研究員
(現:東洋大学食環境科学部食環境科学科 助教)
市橋 泰範 研究当時:チームリーダー
(現:環境資源科学研究センター ホロビオント・レジリエンス研究チームチームディレクター)
環境資源科学研究センター 代謝システム研究チーム
藤 佑志郎 研究員
平井 優美 チームディレクター
(兼:質量分析・顕微鏡解析ユニット ユニットリーダー)
論文情報
雑誌名:Briefings in Bioinformatics
題 名:I-SVVS: Integrative stochastic variational variable selection to explore joint patterns of multi-omics microbiome data
著者名:Tung Dang, Yushiro Fuji, Kie Kumaishi, Erika Usui, Shungo Kobori, Takumi Sato, Megumi Narukawa, Yusuke Toda, Kengo Sakurai, Yuji Yamasaki, Hisashi Tsujimoto, Masami Yokota Hirai, Yasunori Ichihashi, Hiroyoshi Iwata*
DOI: 10.1093/bib/bbaf132
URL: https://academic.oup.com/bib/article-lookup/doi/10.1093/bib/bbaf132
研究助成
本研究は、JSPS科研費「植物-微生物叢相互作用のマルチオミクス階層モデリングとその高速アルゴリズムの開発(課題番号:JP21J21850)」, JST CREST「植物環境応答のモデル化に基づく発展型ゲノミックセレクションシステムの開発(課題番号:JPMJCR16O2)」の支援を受けて行われました。
用語解説
(注1)クラスタリング(Clustering):
データをその特徴に基づいて、似たもの同士を同じグループ(クラスタ)に分類する手法。同じクラスタのデータは互いに類似し、異なるクラスタ間では異なるように分ける。
(注2)ベイズ非パラメトリック手法:
ベイズ統計の枠組みで、モデルの複雑さ(パラメータ数)をあらかじめ固定せず、データに応じて柔軟に調整する手法。複雑な構造をもつデータの表現に適している。
(注3)変分推論(Variational Inference):
計算が困難なベイズ推論の事後分布を、より単純な分布で近似する手法。具体的には、事後分布に近い単純な分布を仮定し、その近似の精度を表す指標を最大化することで、元の複雑な問題を扱いやすくする。確率的変分推論(Stochastic Variational Inference: SVI)では、全データを使わず一部(ミニバッチ)を用いて効率的に最適化を行うことで、大規模データにも対応可能となる。
(注4)無限ディリクレ多項分布混合モデル:
データを多項分布でモデル化し、クラスタ数をあらかじめ固定せず、データから最適な数を自動的に推定するクラスタリング手法。ディリクレ過程を用いて、データの構造に応じた最適なクラスタ数を学習する。
(注5)無限ガウス混合モデル:
ガウス分布の混合でデータを表現しつつ、必要に応じてクラスタ数(成分数)を増減させるベイズ非パラメトリック手法。ディリクレ過程により、データ構造に適した数のクラスタを学習する
(注6)iClusterPlus:
遺伝子発現やDNAメチル化など複数のオミクスデータを統合し、潜在変数モデルに基づいてサンプルを同時にクラスタリングする手法。異なるデータタイプの情報を組み合わせることで、より精度の高いサブタイプ分類が可能となる。
(注7)Clusternomics:
異種オミクスデータ(例:遺伝子発現、メチル化、プロテオミクスなど)を統合的にクラスタリングするベイズ的手法。各データセットに特有の構造を保持しつつ、共通するクラスタ構造も同時に推定する。
問合せ先
(研究内容については発表者にお問合せください)
東京大学 大学院農学生命科学研究科 生産・環境生物学専攻
教授 岩田 洋佳(いわた ひろよし)
Tel:03-5841-5069 E-mail:hiroiwata[at]g.ecc.u-tokyo.ac.jp
〈報道に関する問合せ〉
東京大学 大学院農学生命科学研究科・農学部
事務部 総務課総務チーム 総務・広報情報担当(広報情報担当)
Tel:03-5841-8179,5484 FAX:03-5841-5028 E-mail:koho.a[at]gs.mail.u-tokyo.ac.jp
理化学研究所 広報部 報道担当
Tel:050-3495-0247 E-mail:ex-press[at]ml.riken.jp
※上記の[at]は@に置き換えてください。